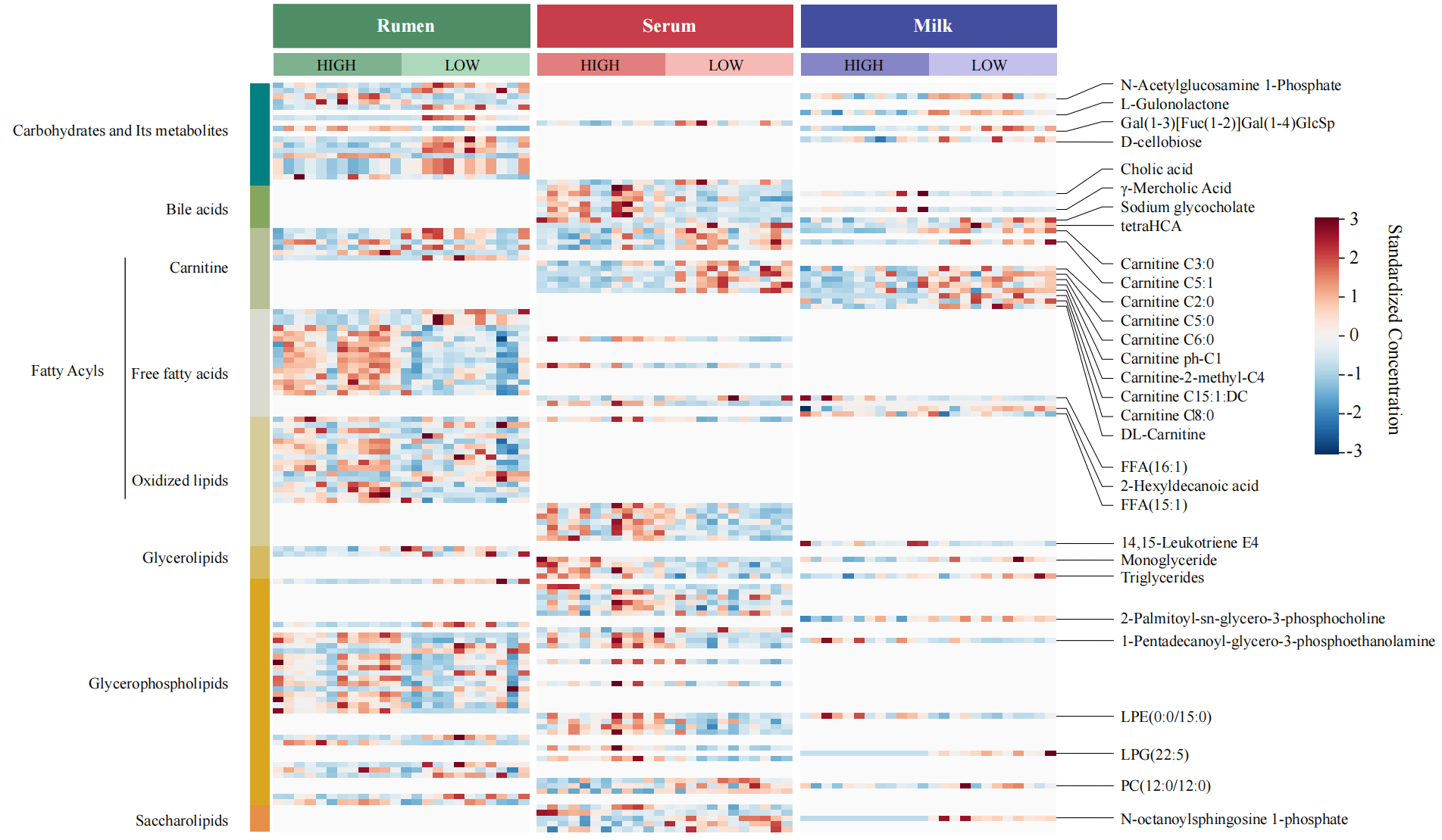
多部位样本联合代谢组学共有与特有代谢产物组合热图
写在前面
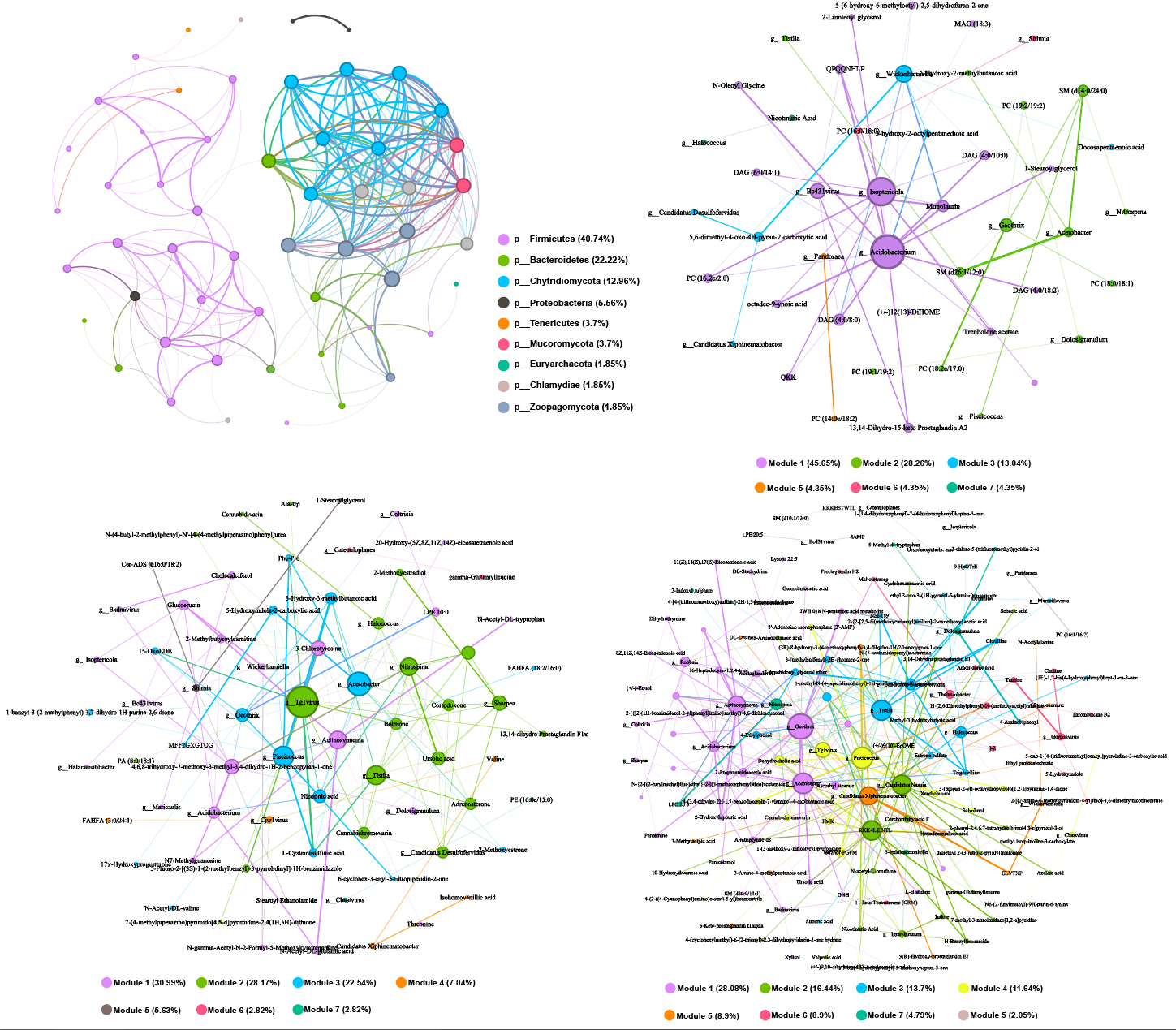
拿到了瘤胃液、血液、牛奶的代谢组学结果,想找出这几种体液之间差异代谢物的关联,偶然在师妹那里看到了下面这个图,描述的是两组间差异脂质类代谢物在三种体液内的分布与代谢组鉴定强度,用这样一种组合热图的方式展现,包含了每种体液包含差异代谢物的种类、差异代谢物在组间的鉴定强度、差异代谢物的分类,以及能看出共有、特有以及流向分布等信息,不只是代谢组学,同理可运用到其他多组织部位的联合组学。下面用我自己的数据描述复刻过程。
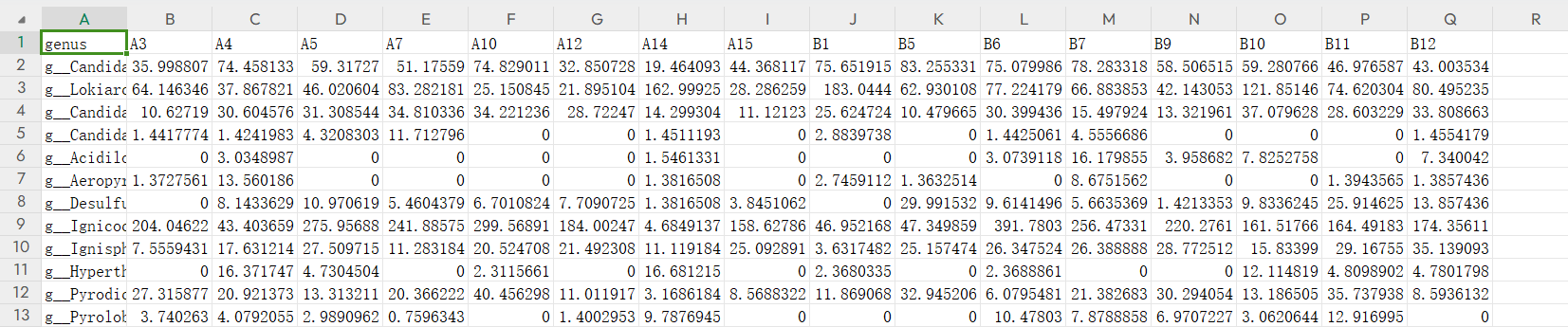
拿到多组学的数据后一直在找合适的方法将二者进行关联,比如我这里是三种体液的代谢组与一种体液的宏基因组。需求是对多组学进行关联分析,直到最近看到不少文章里利用Gephi将相关性表格进行可视化的图,效果还不错,于是写个过程记录自用。这里演示的是属水平的差异菌群相对丰度(宏基因组结果)与代谢组鉴定到的差异代谢物进行关联。
主要分为两个部分:
graphml格式文件主要就是两个需要关联的表格
差异代谢物表格
差异菌属的相对丰度表格
这里注意:原文件如果是新建的excel,默认有3个sheet,一定记得只保留一个sheet后另存为制表符分隔的txt文件,这一点很重要,不然下面分析的时候会从读取就开始报错!
用到Hmisc包和igraph包
这里就直接放代码吧,遇到相关的报错先仔细检查数据,一般是数据格式和内容本身的问题。不行就上网找解决办法。
1 | ###微生物和代谢物的相关性网络 |
plot包出的图
下载地址:The Open Graph Viz Platform,支持中文
可参考 B站:Gephi可视化进阶:教你绘制一个漂亮的网络图
进一步对上面生成的network.graphml文件用Gephi可视化
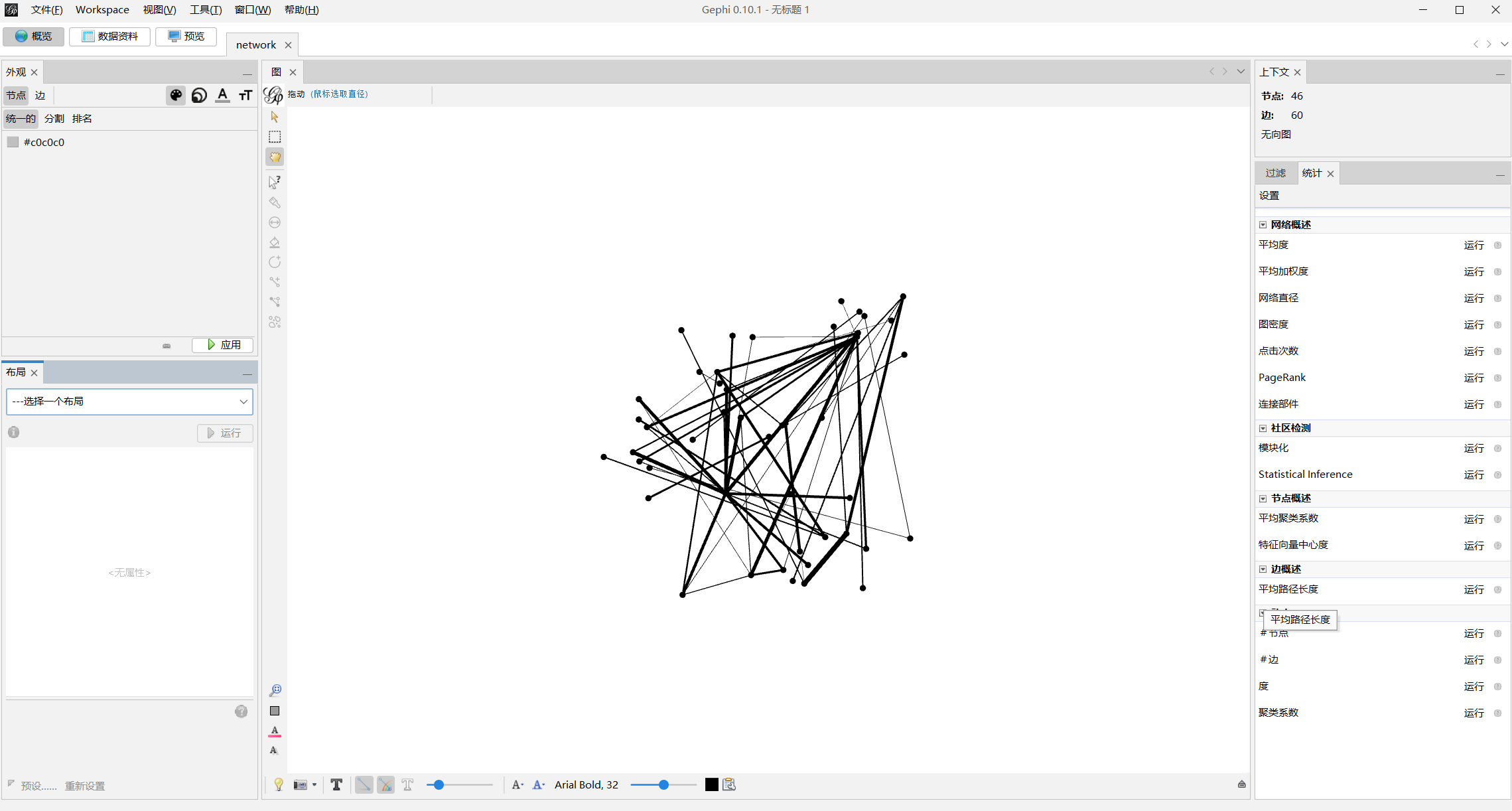
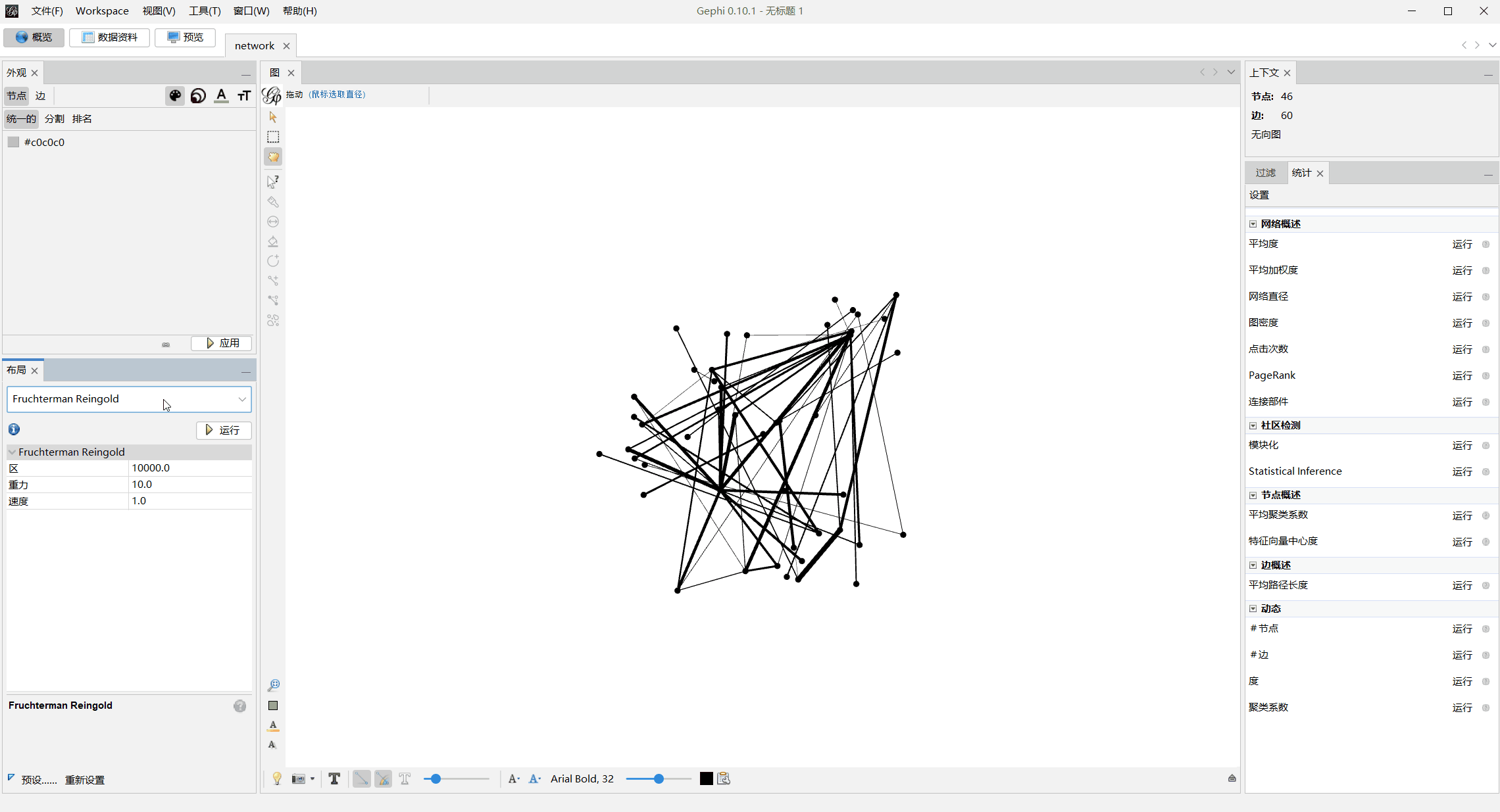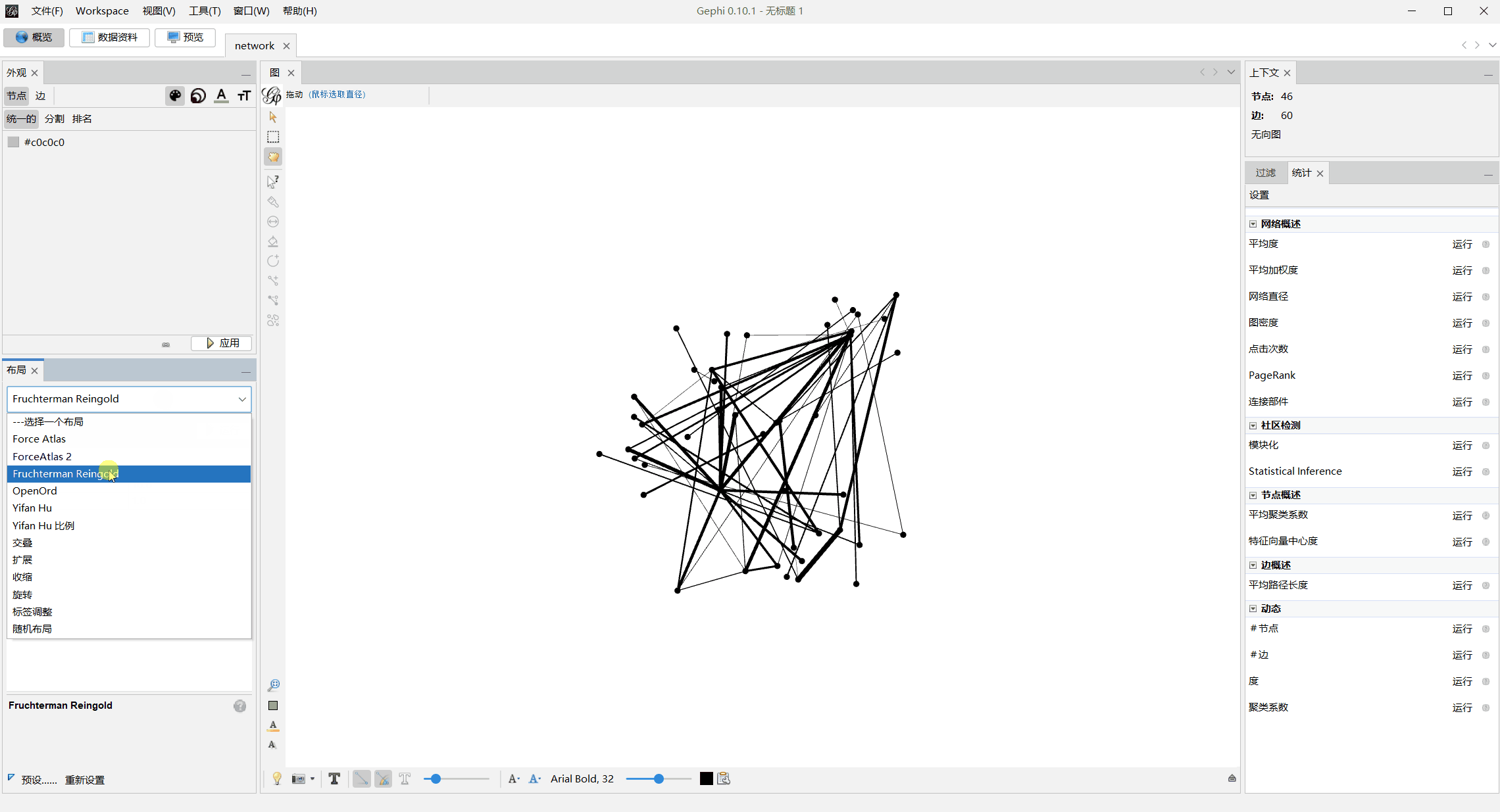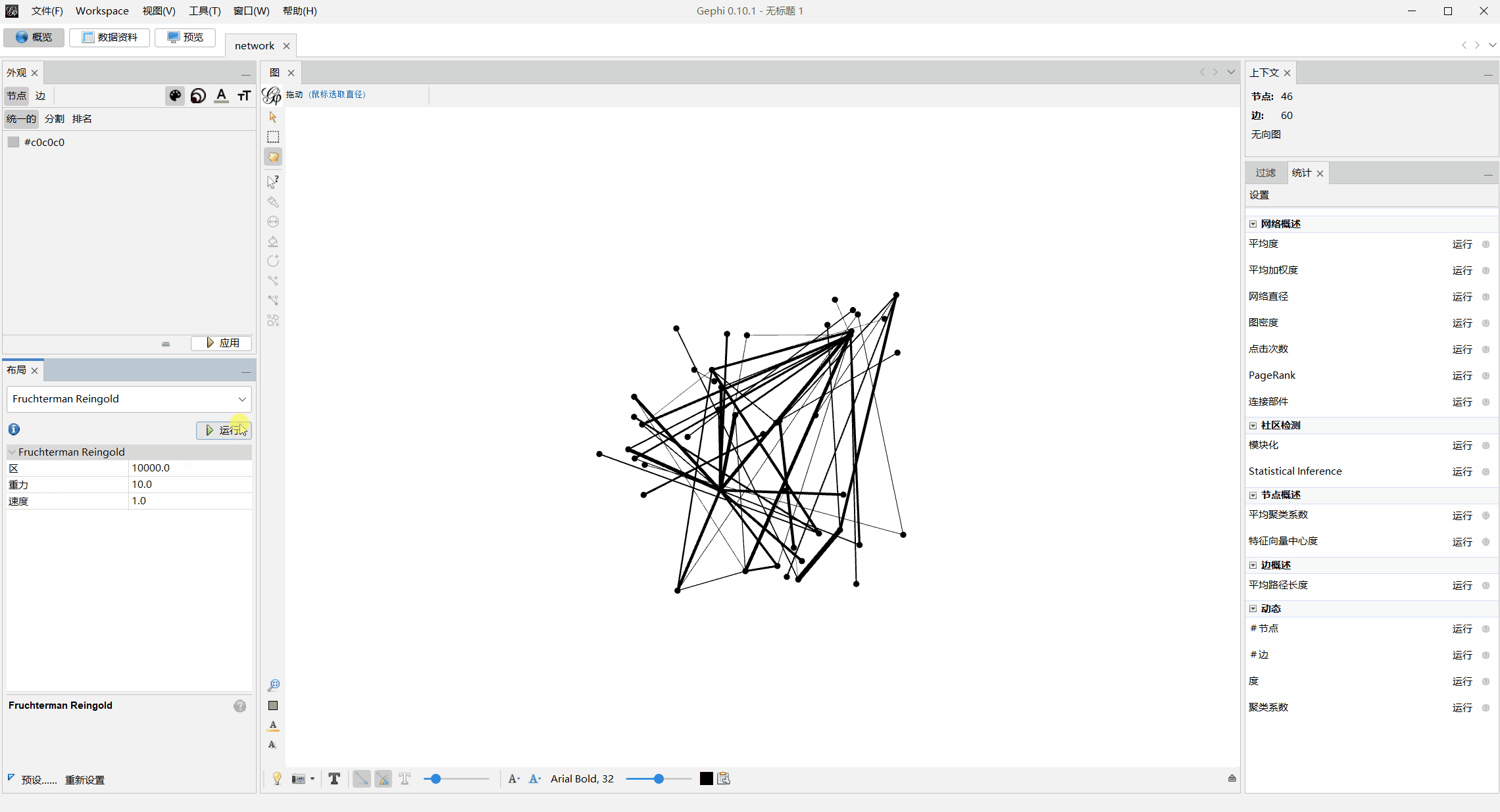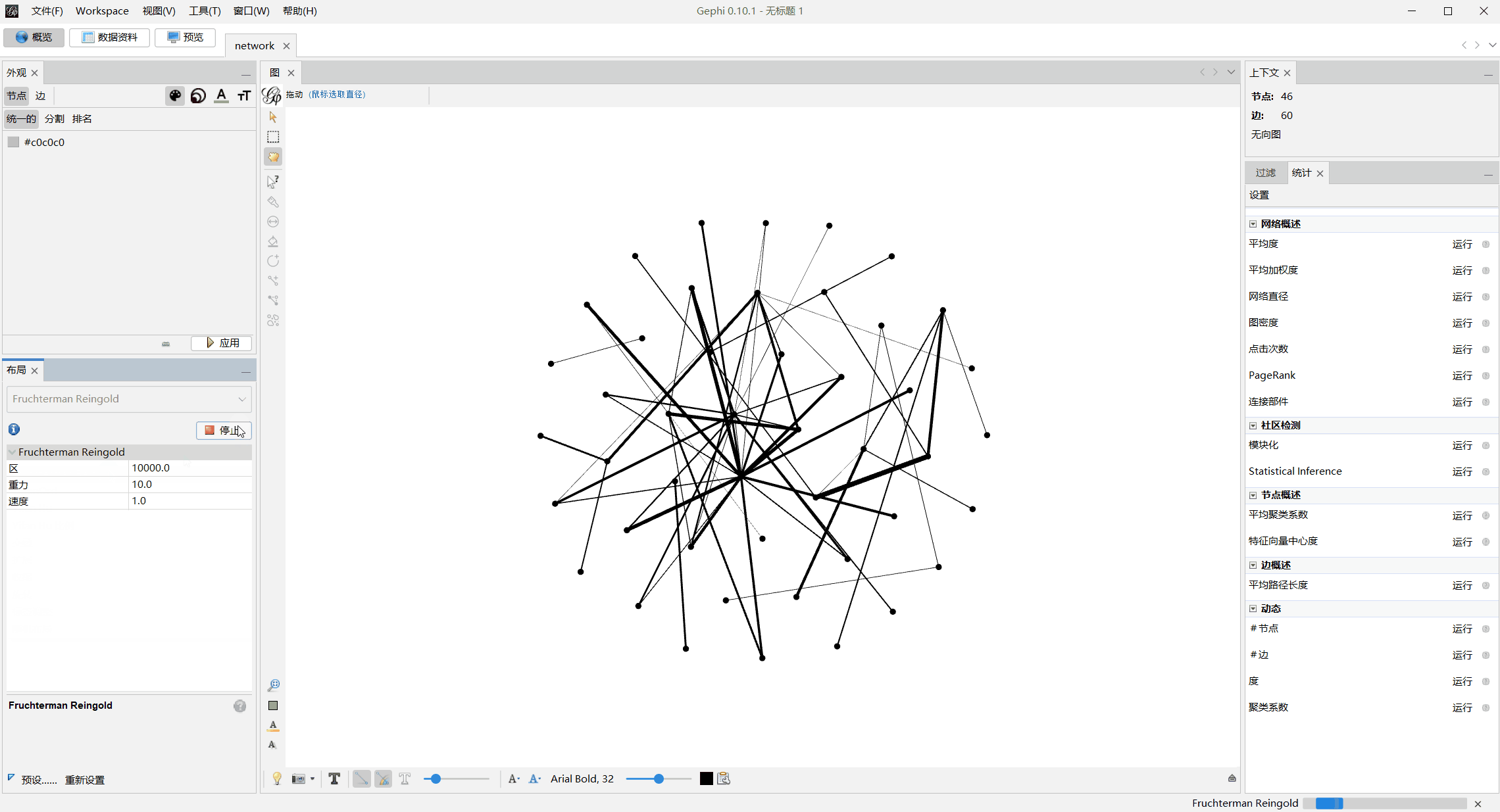
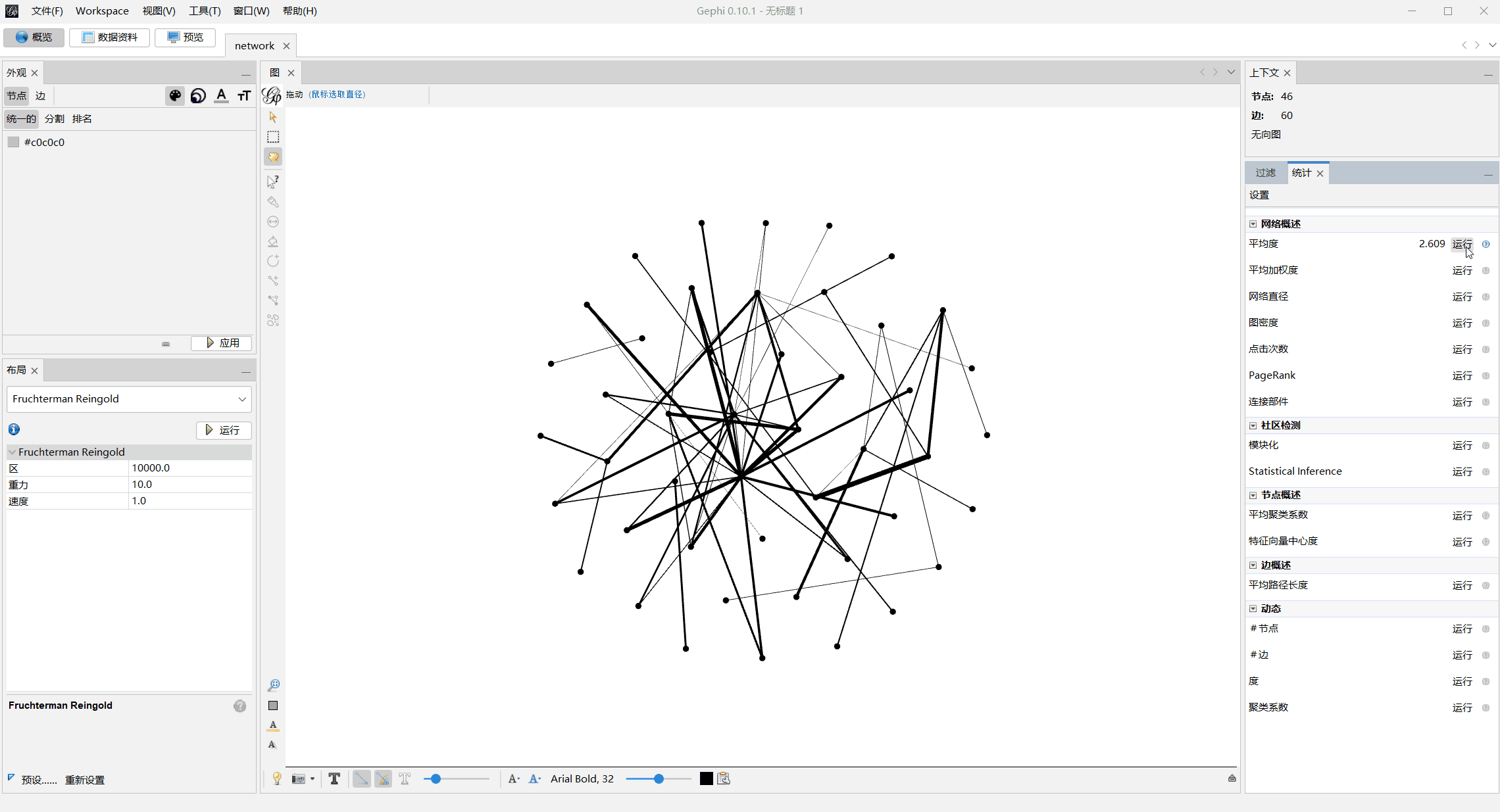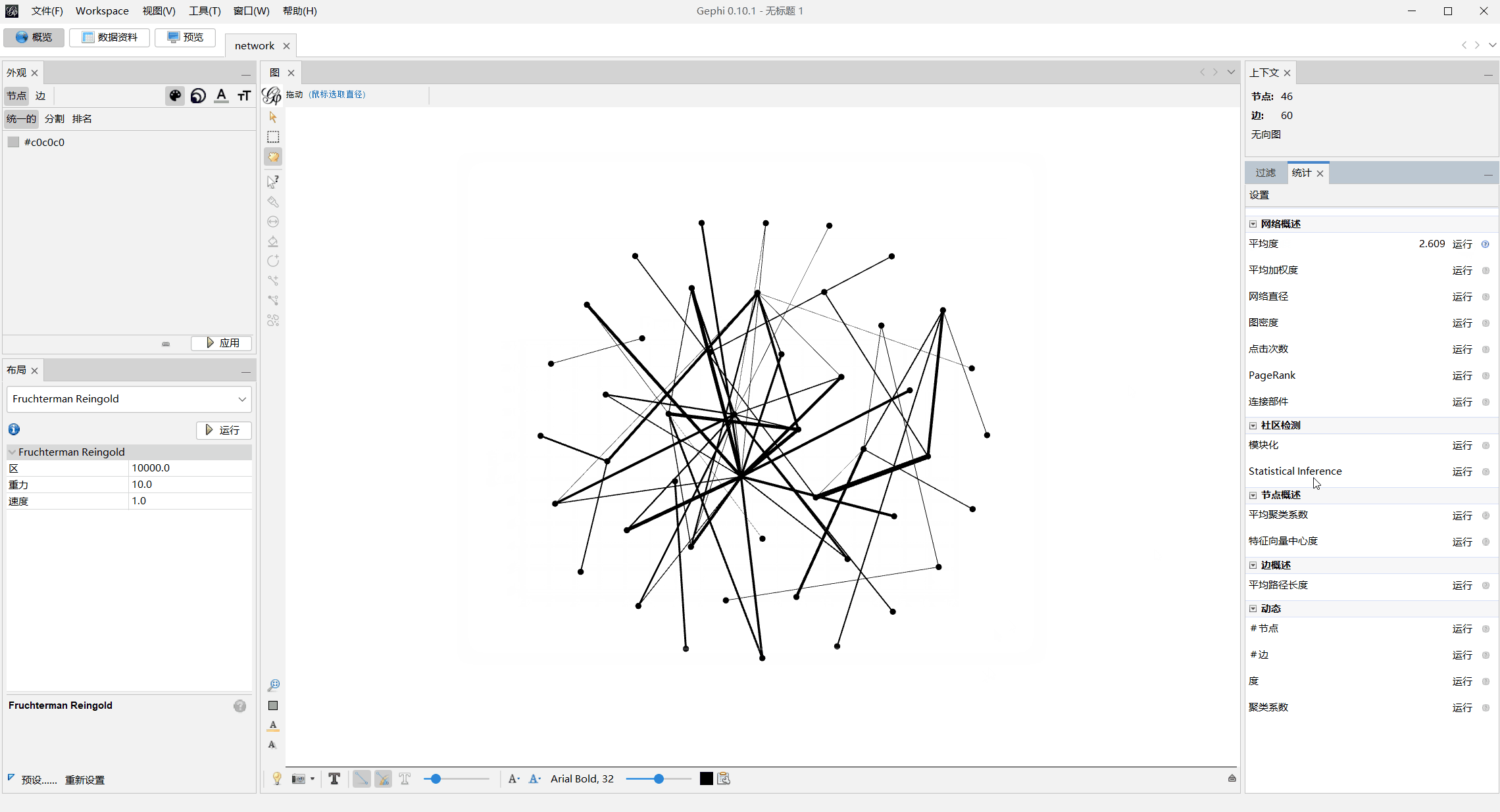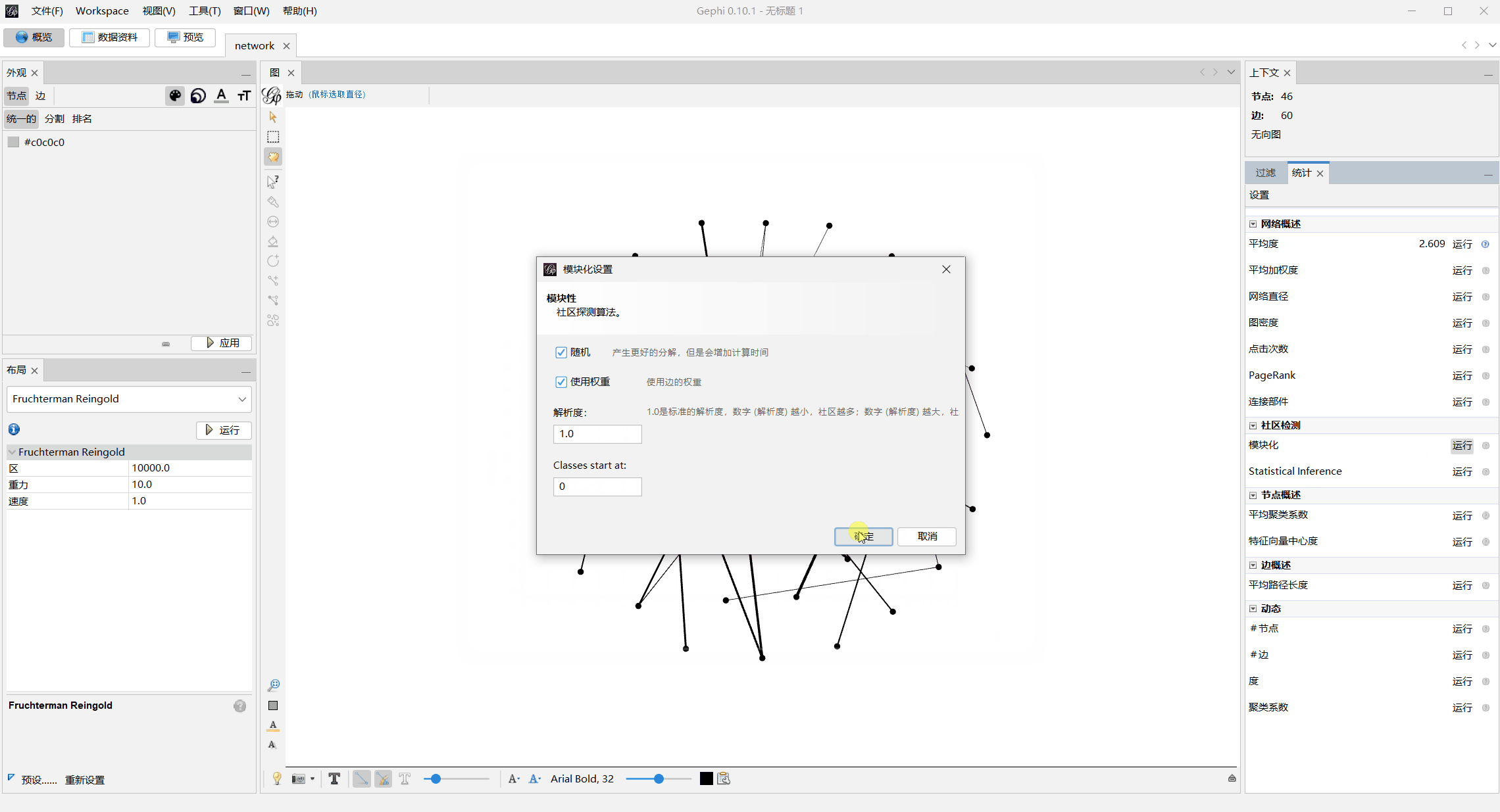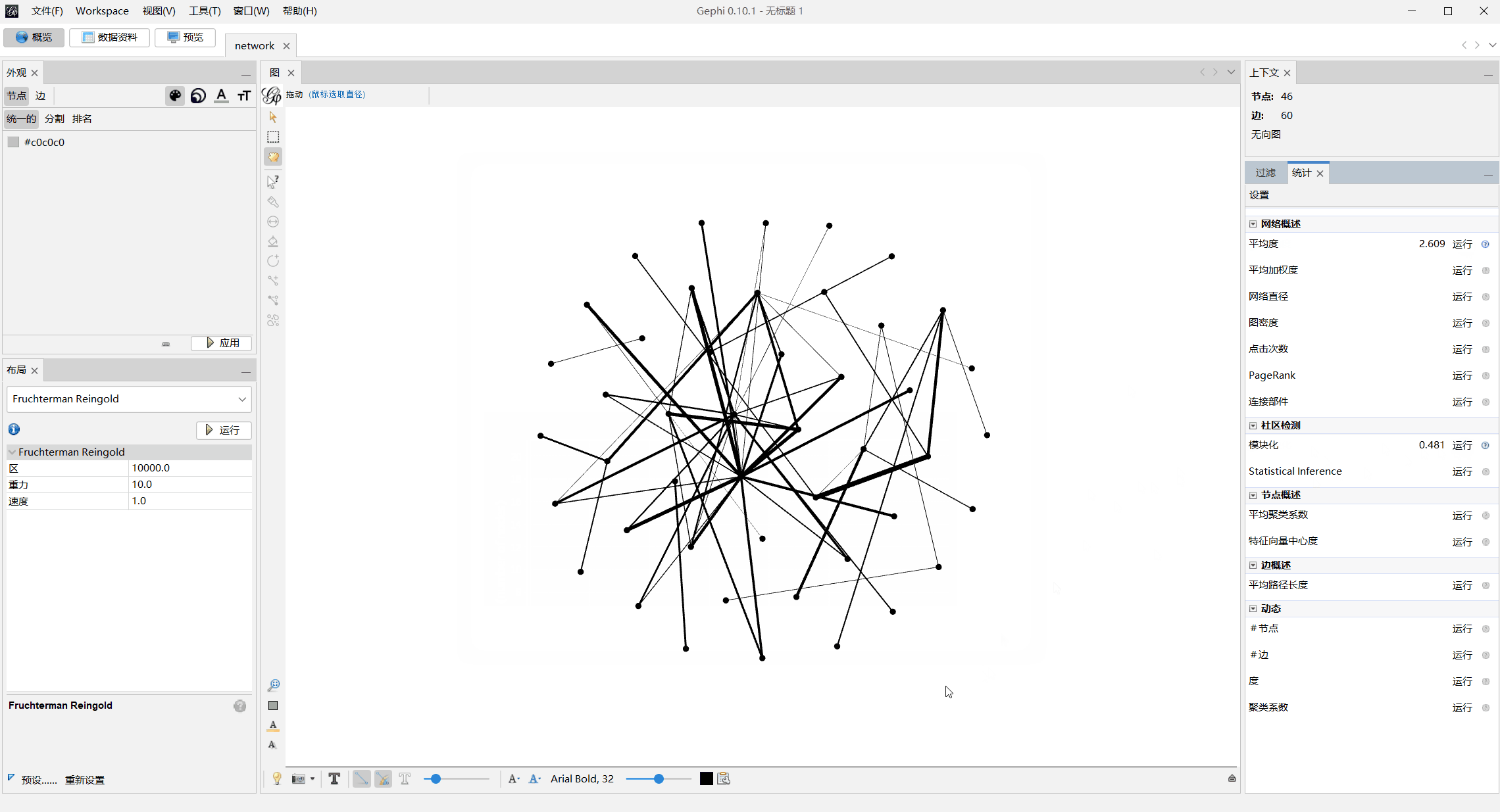
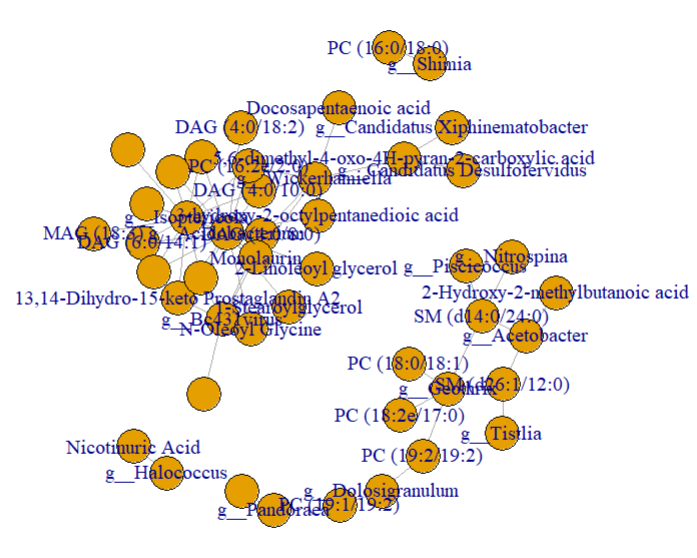
模块化上色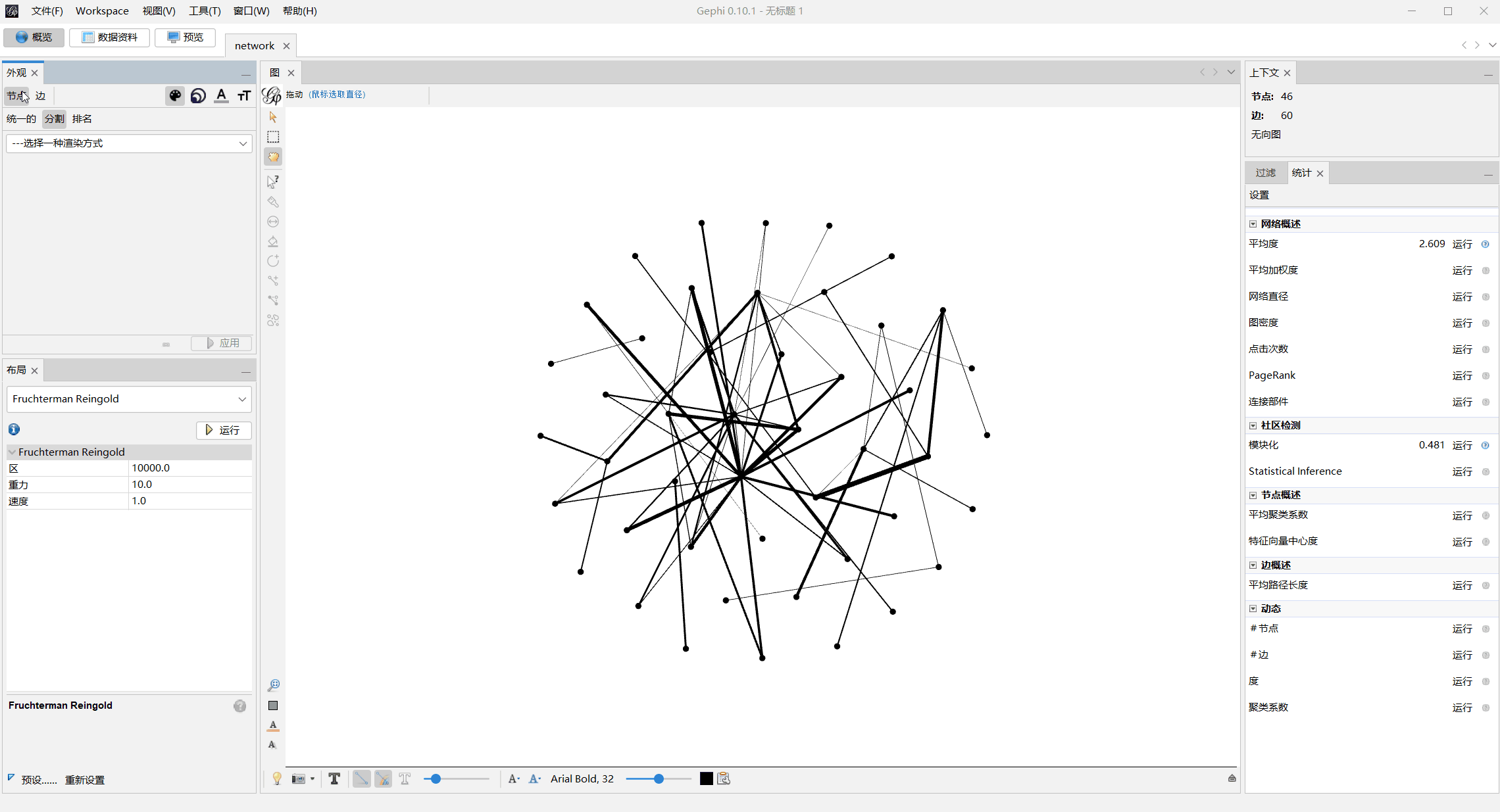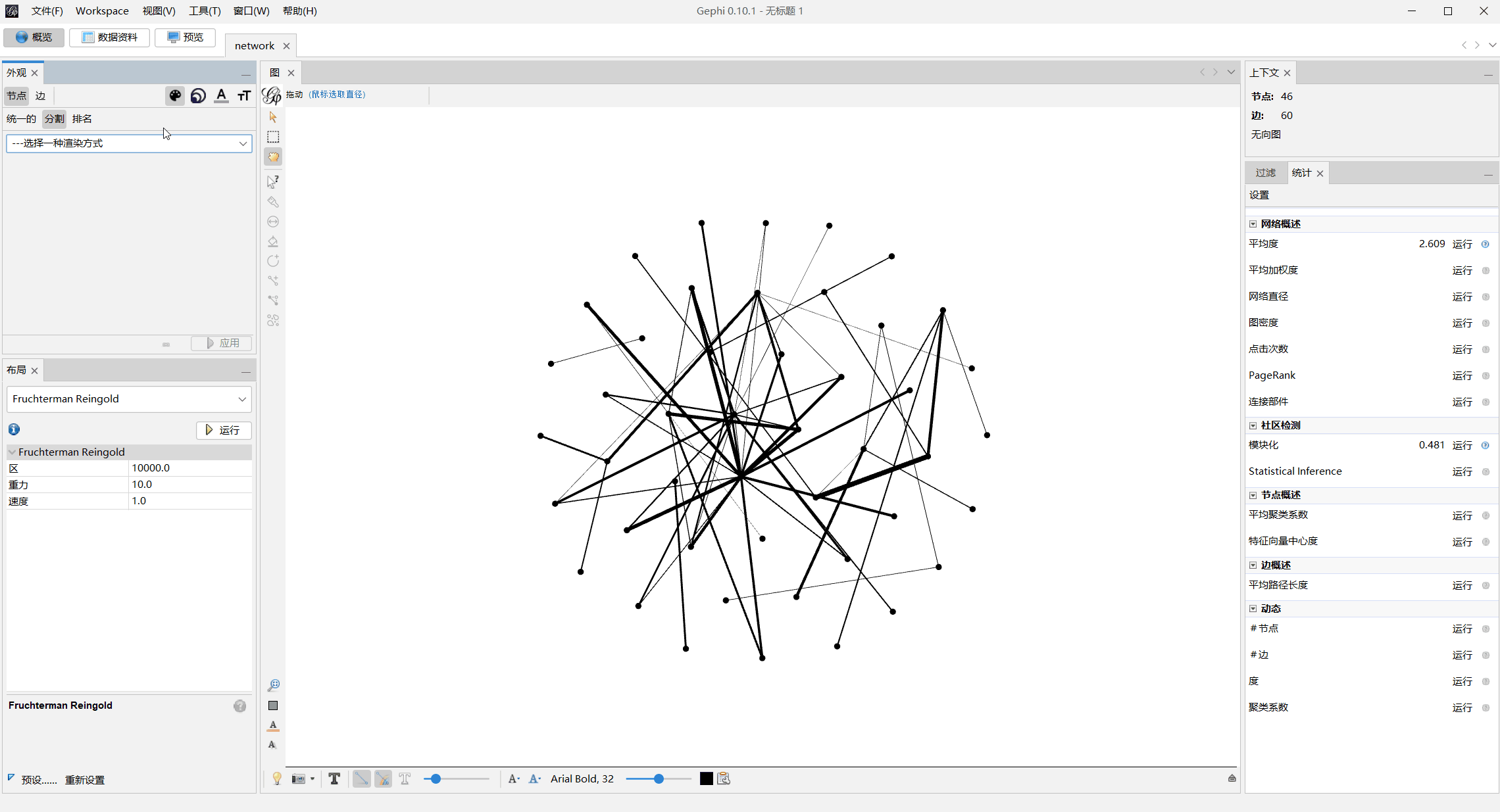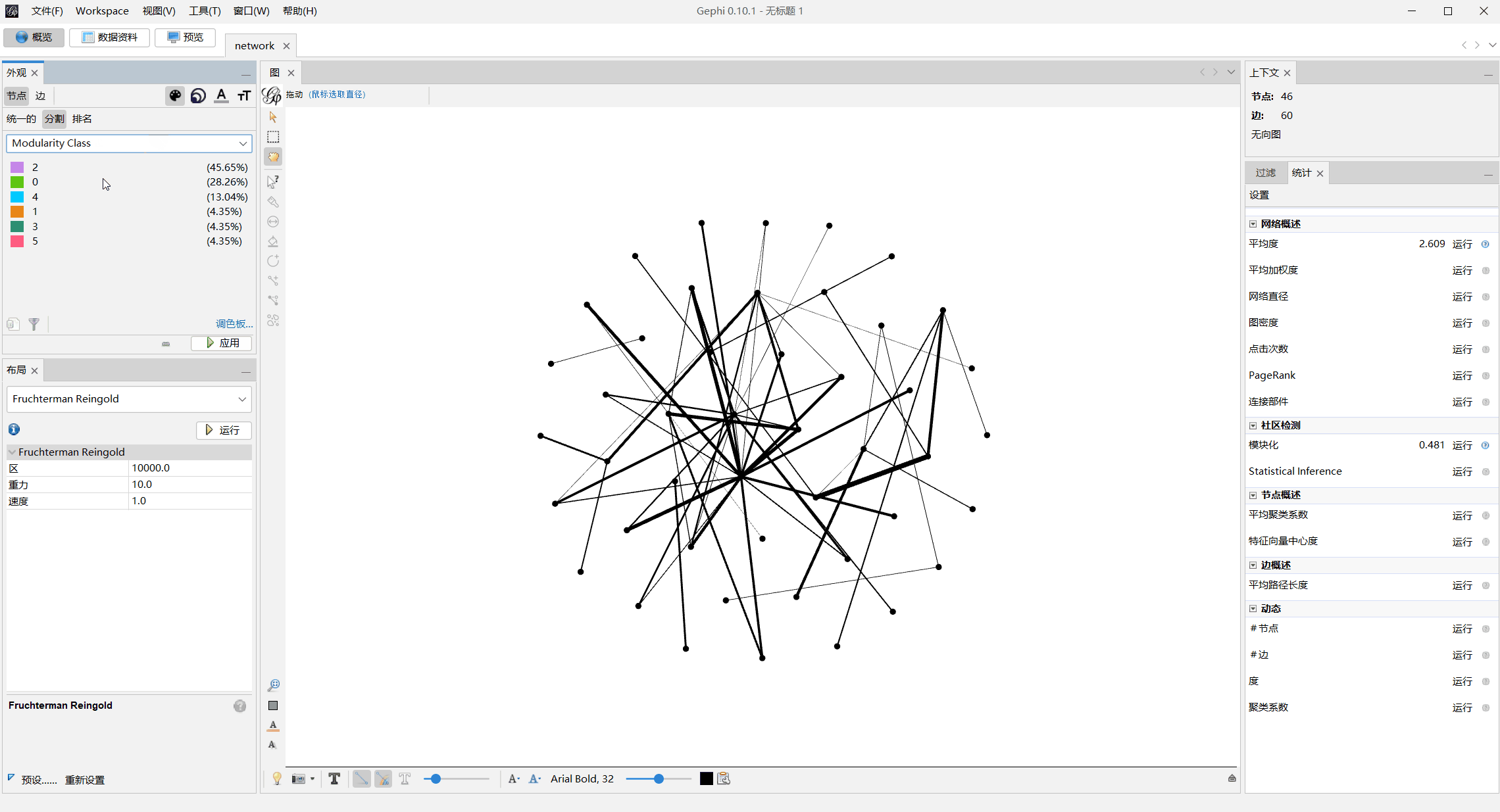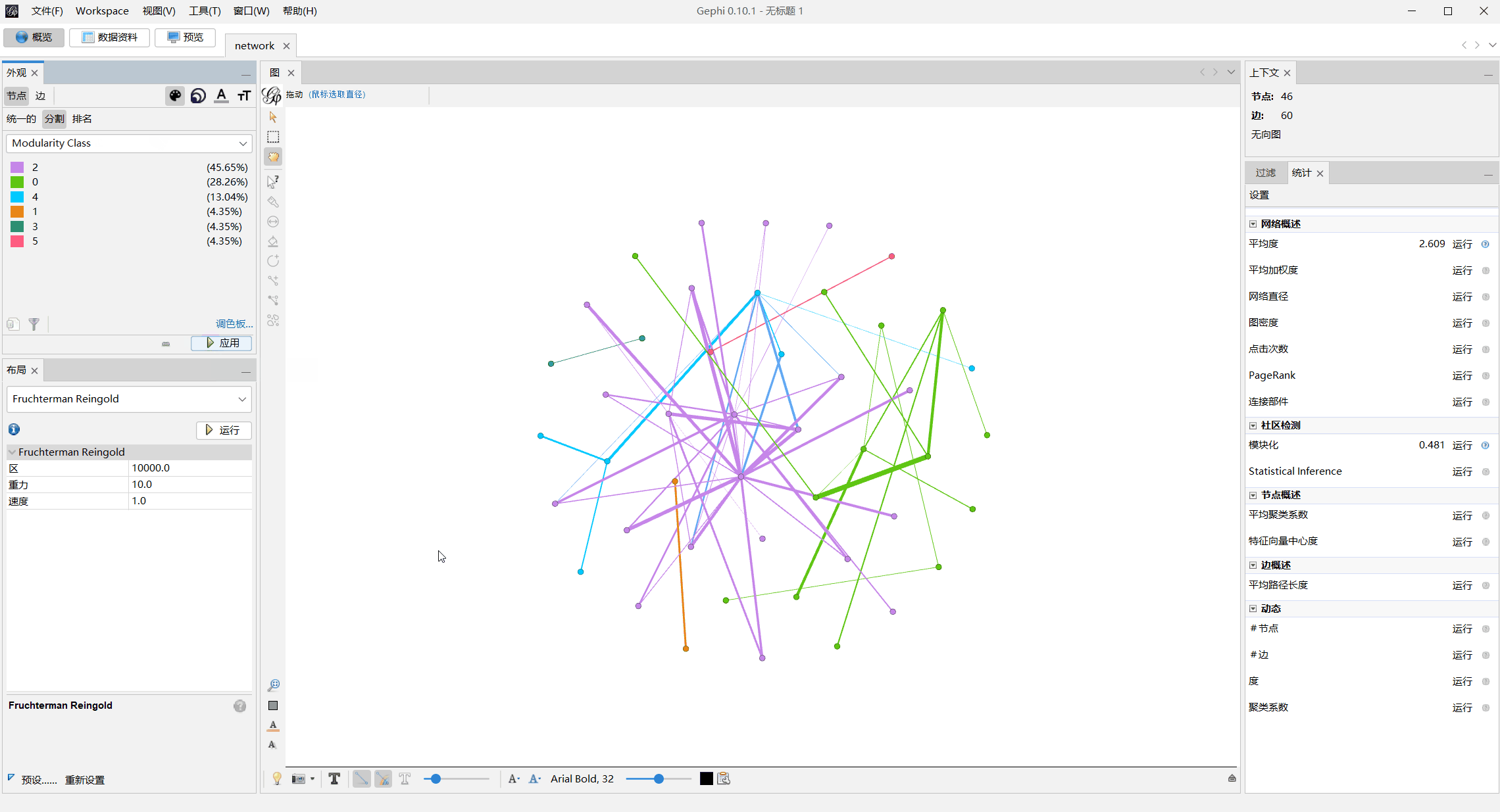
调整节点大小
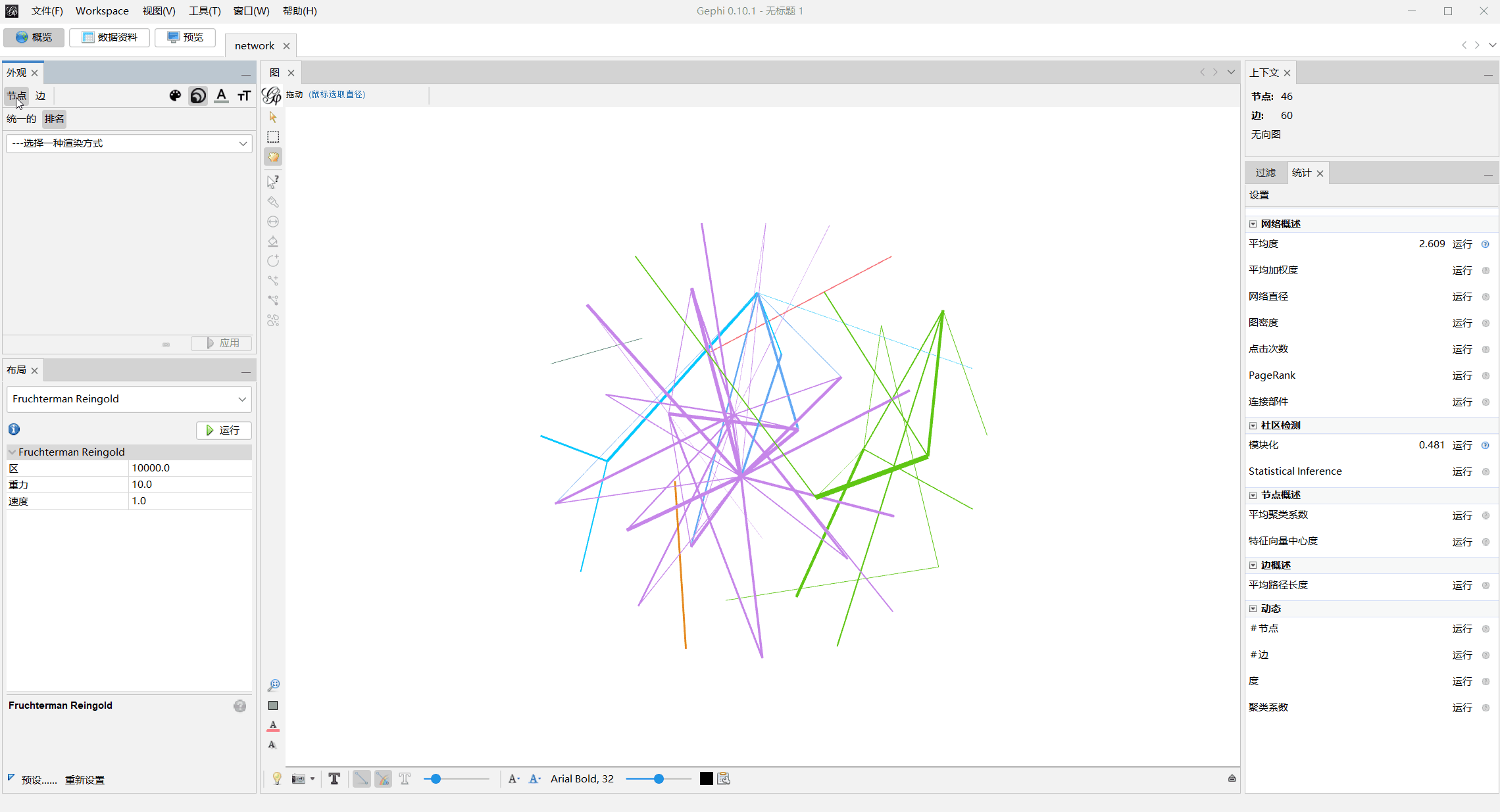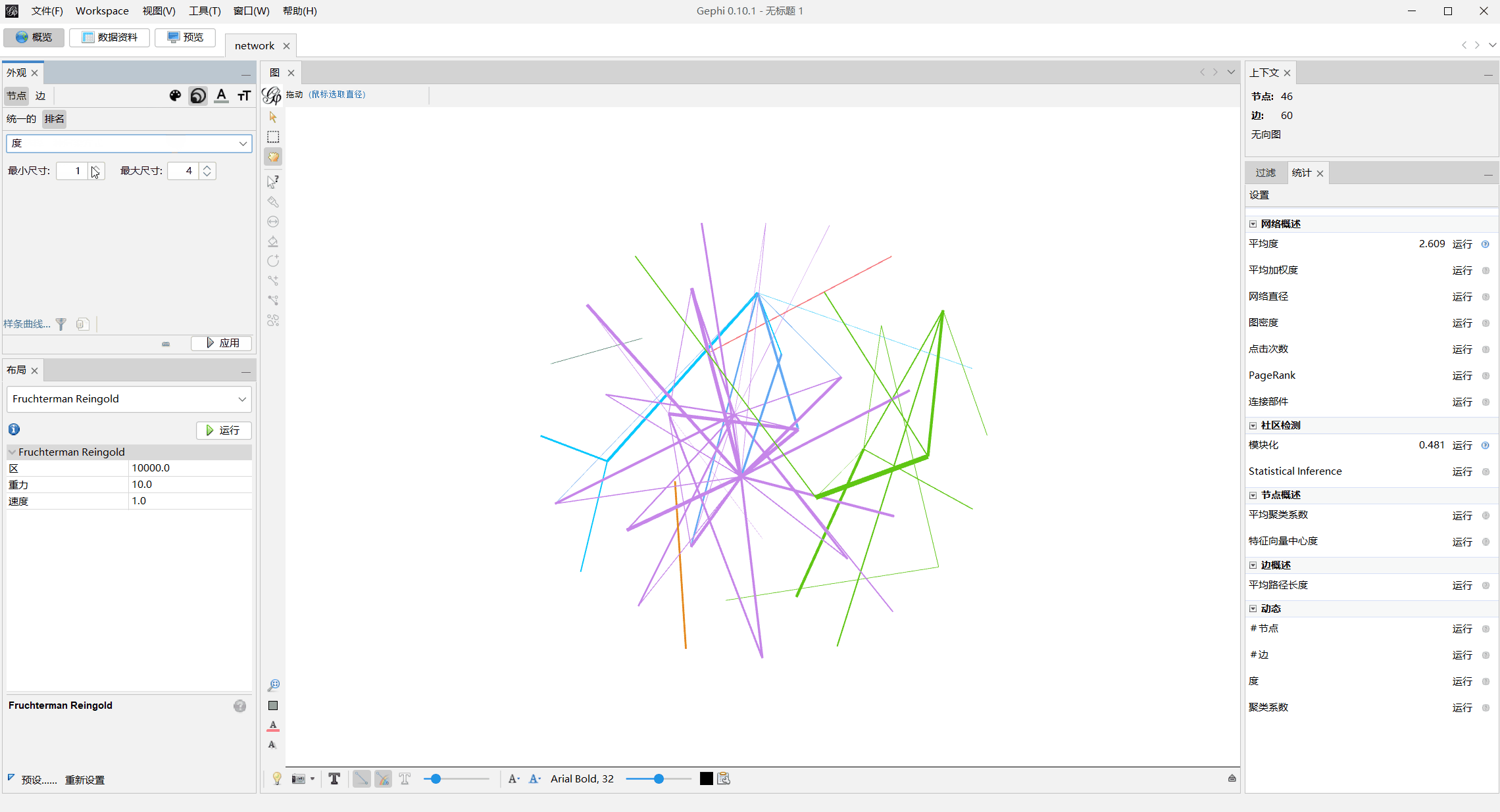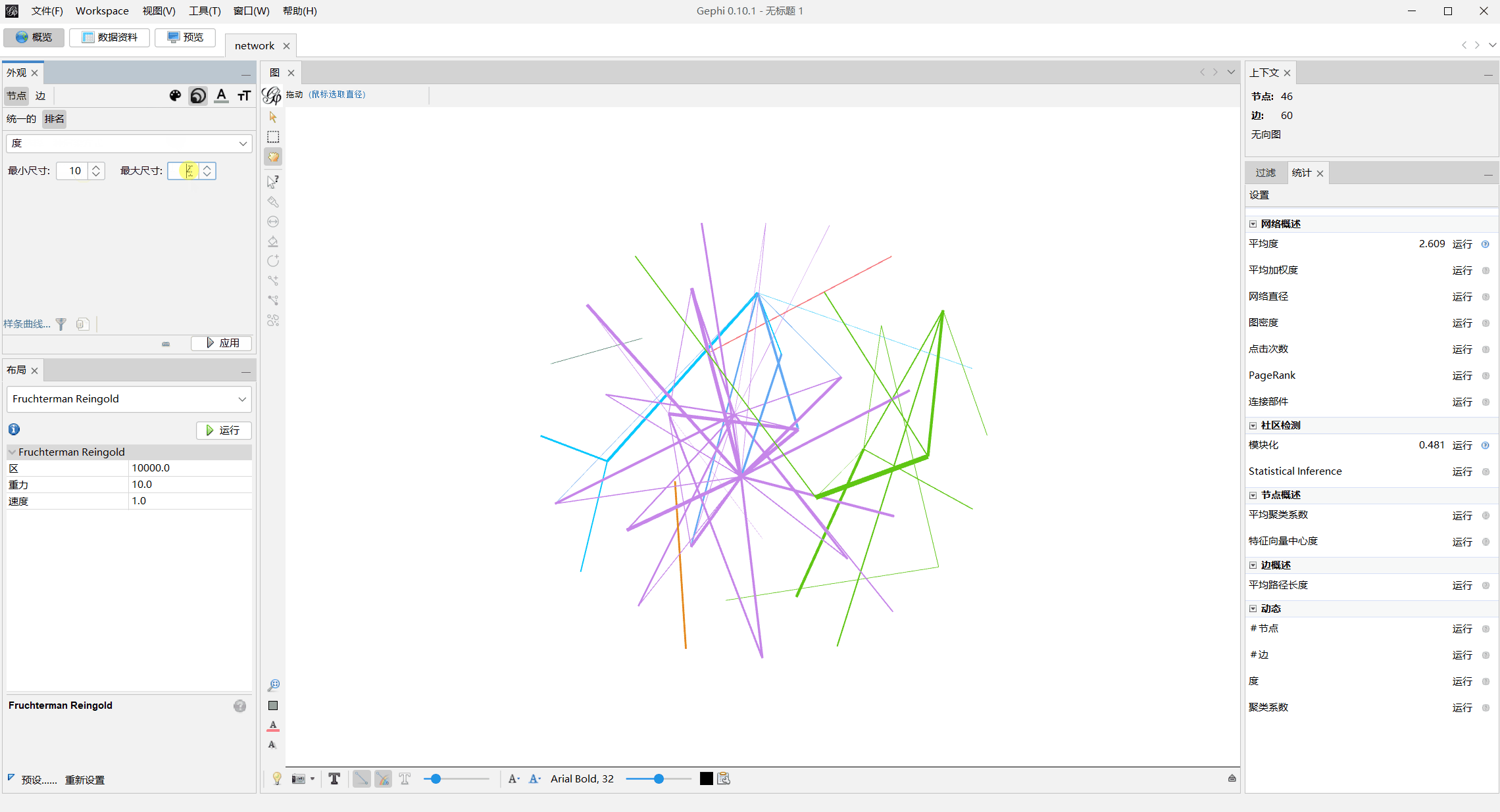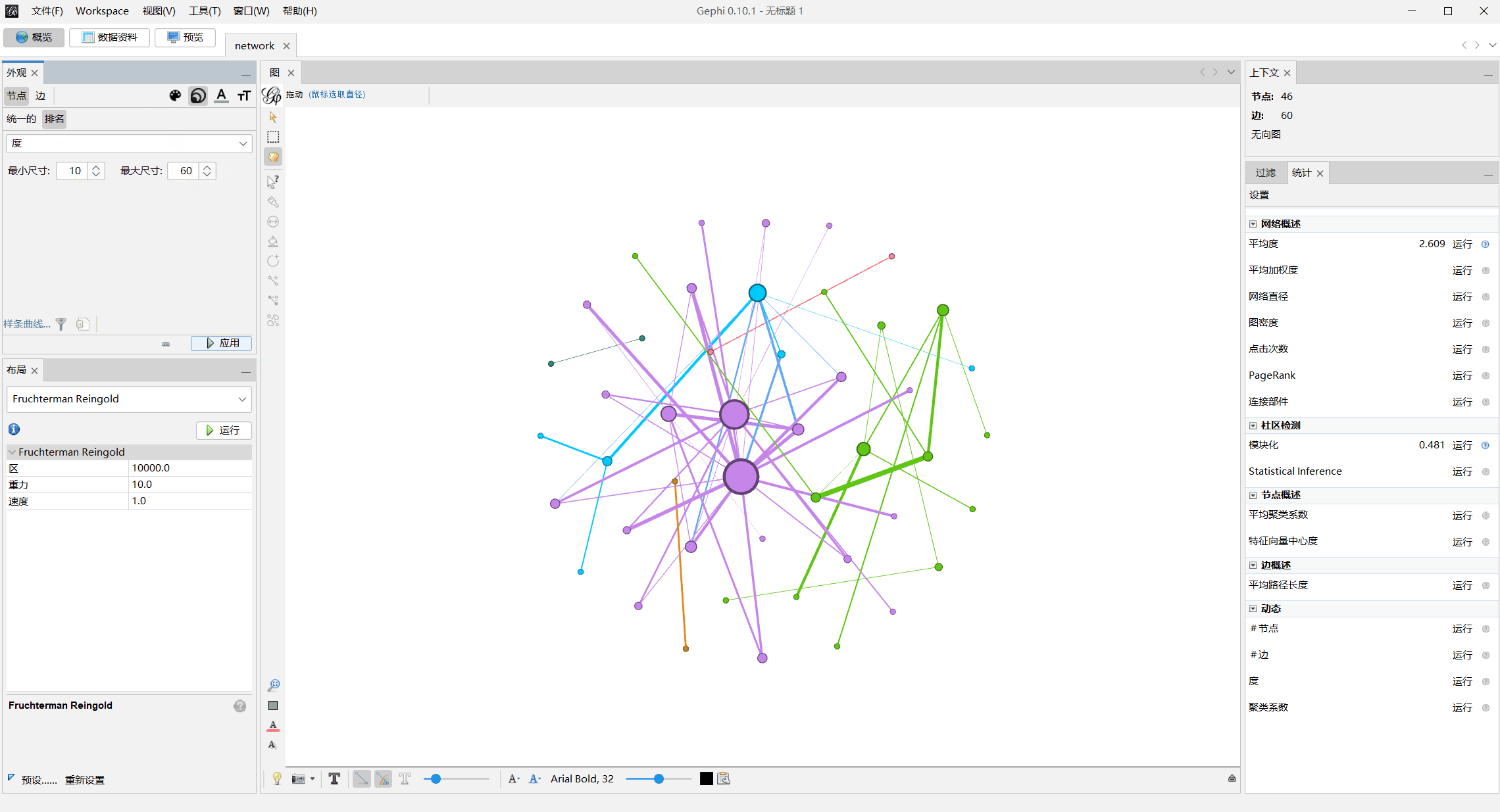
数据资料处替换节点名字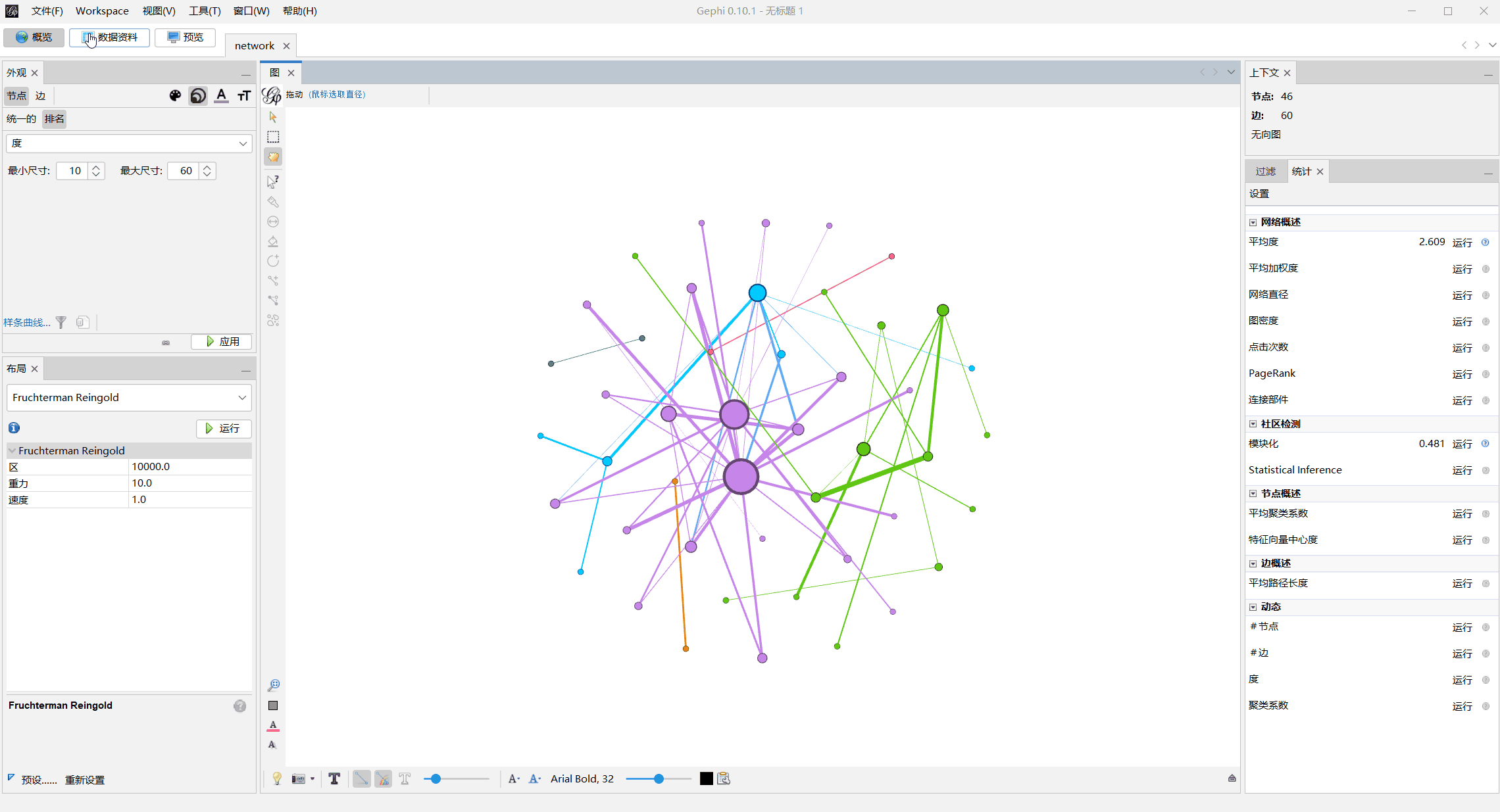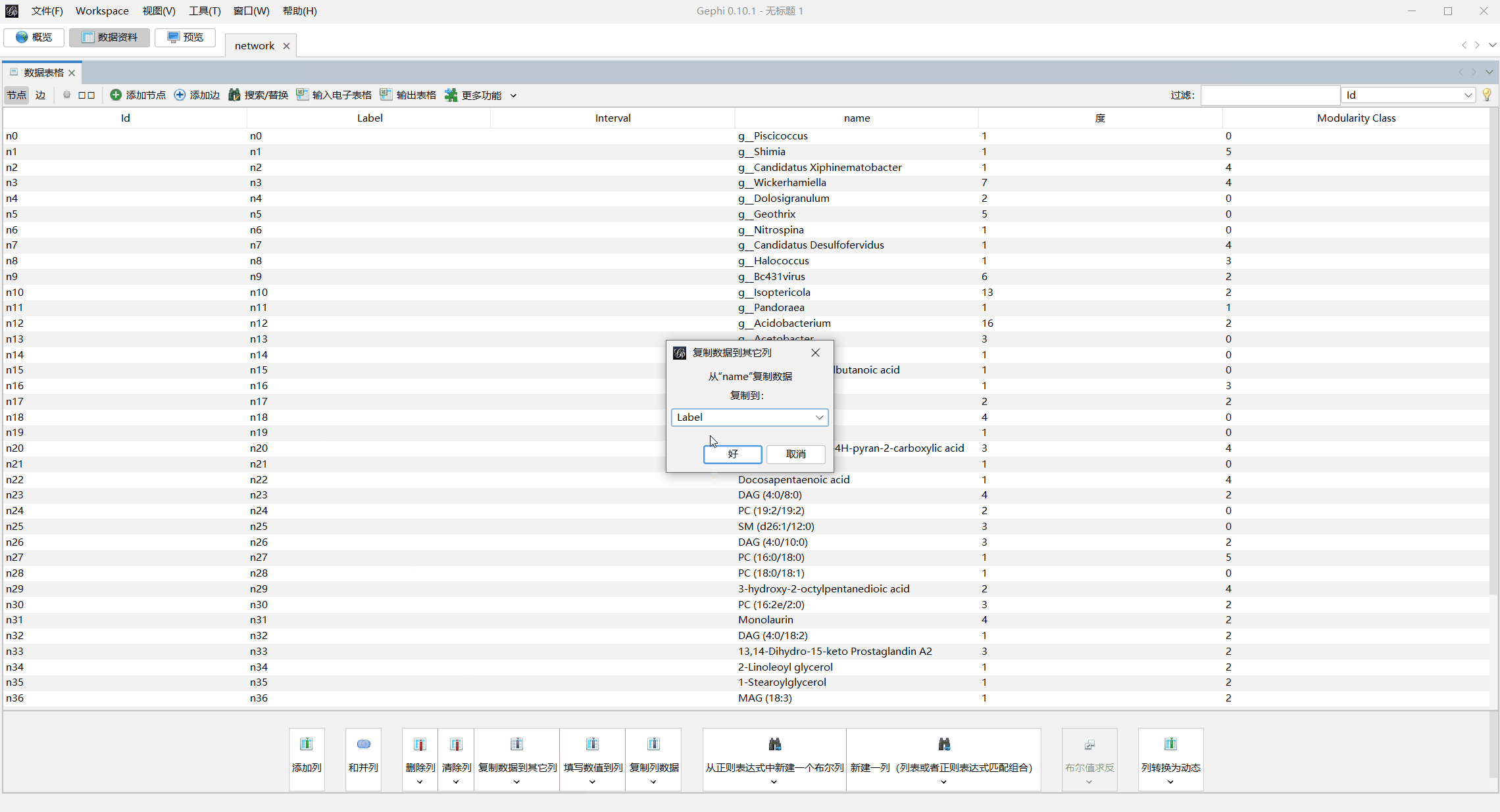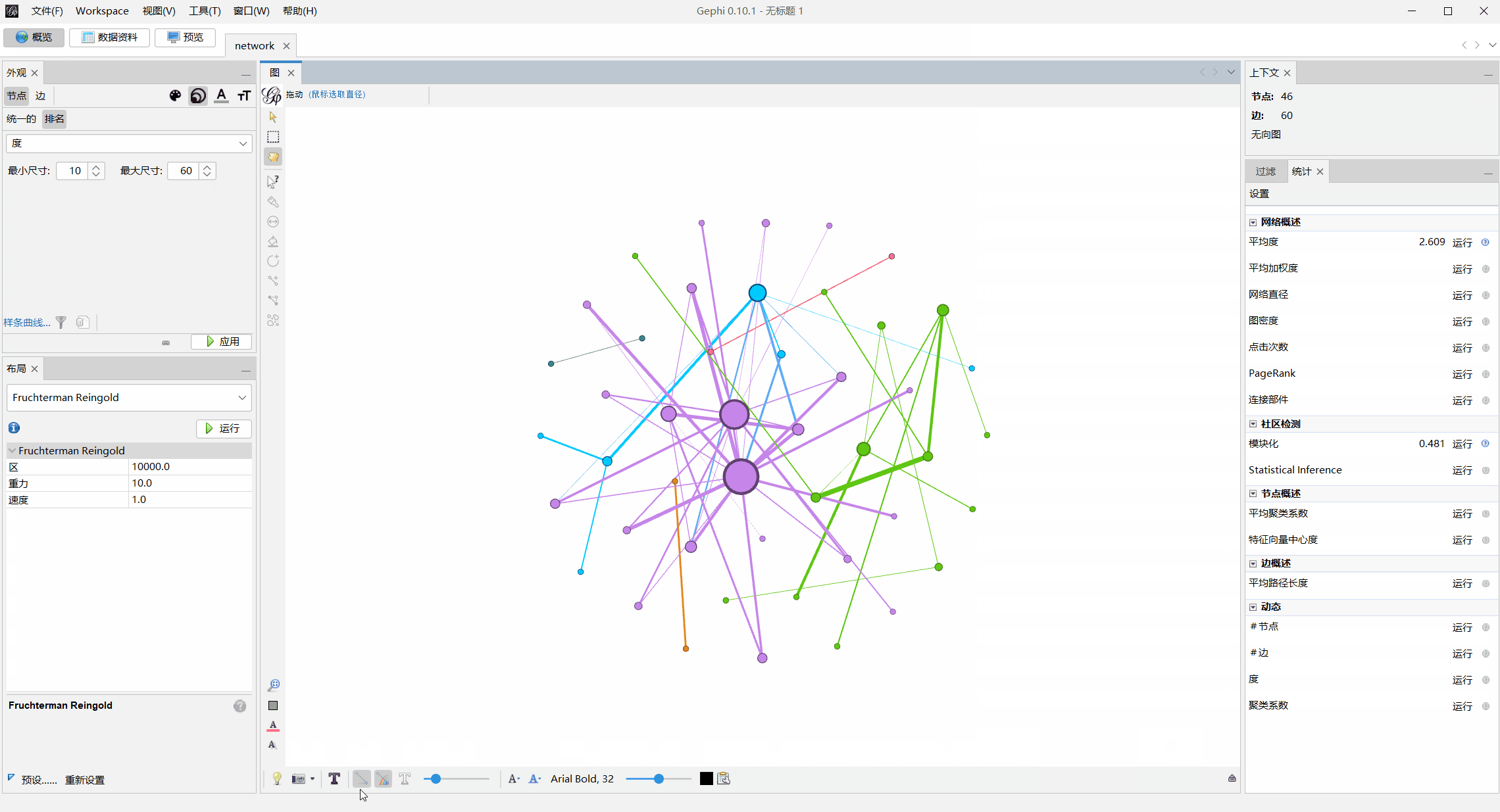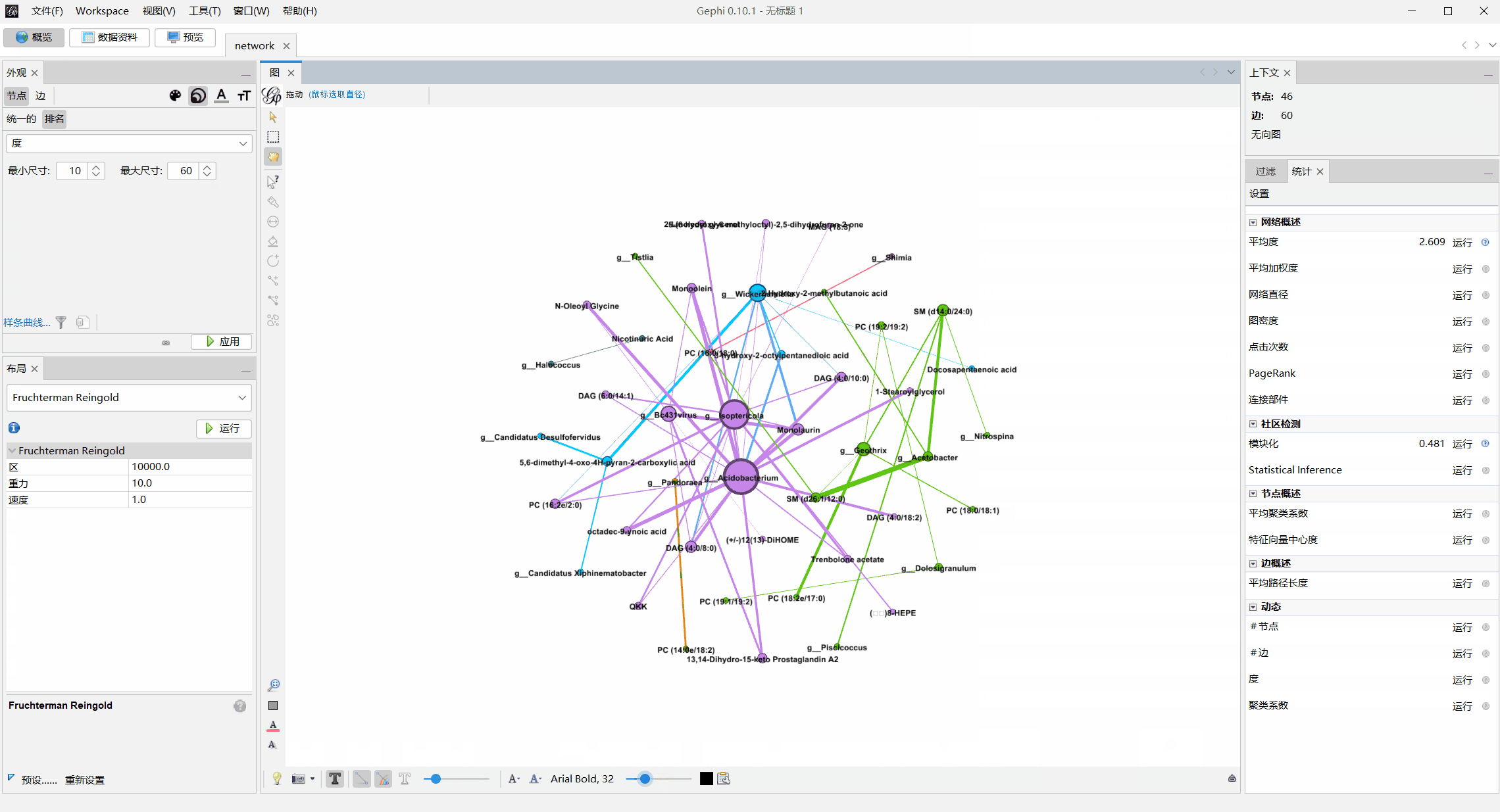
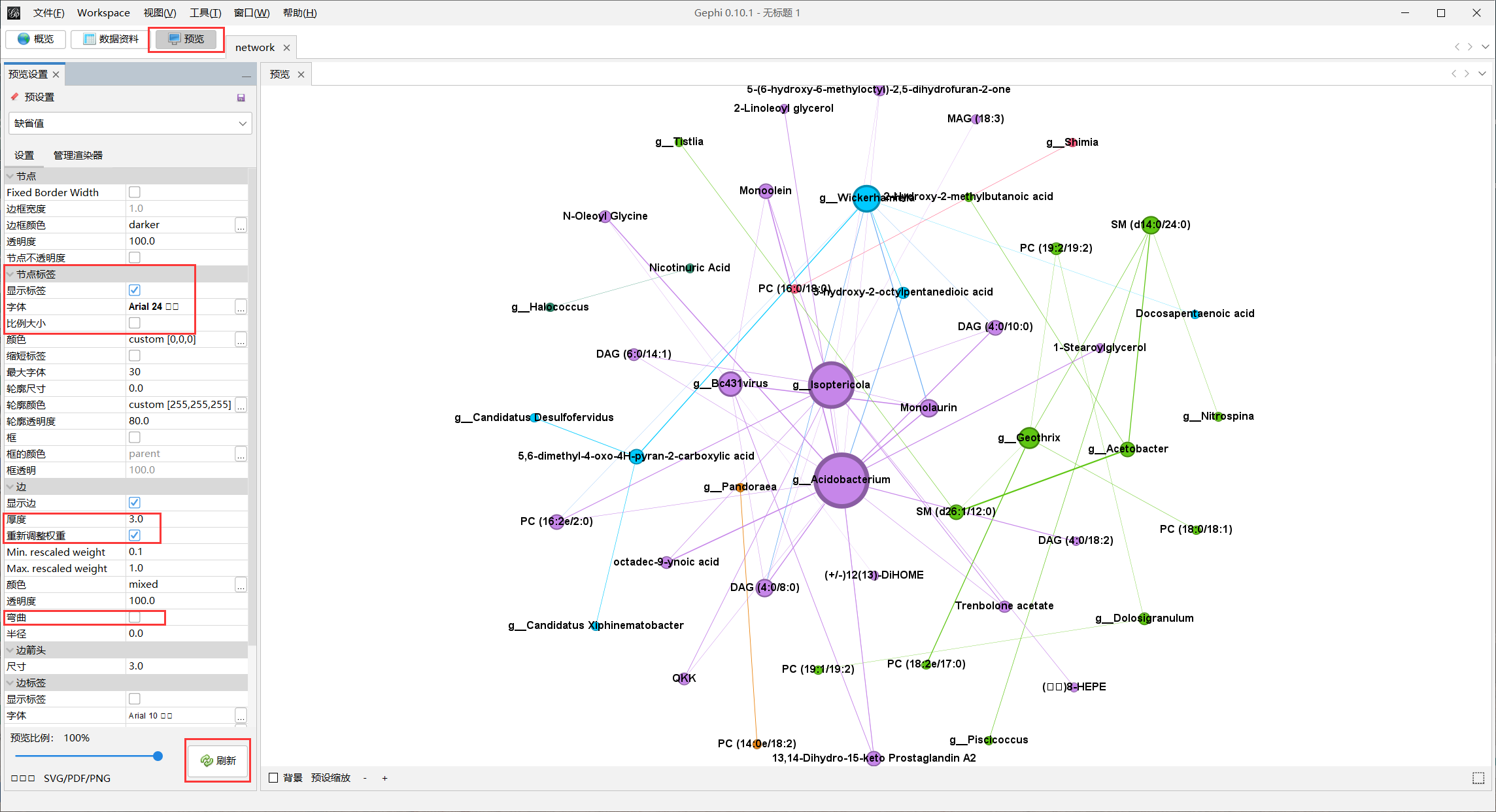
结果图细节修改:在AI中嵌入上面导出的pdf格式结果后补充图例细节即可,下方是效果展示
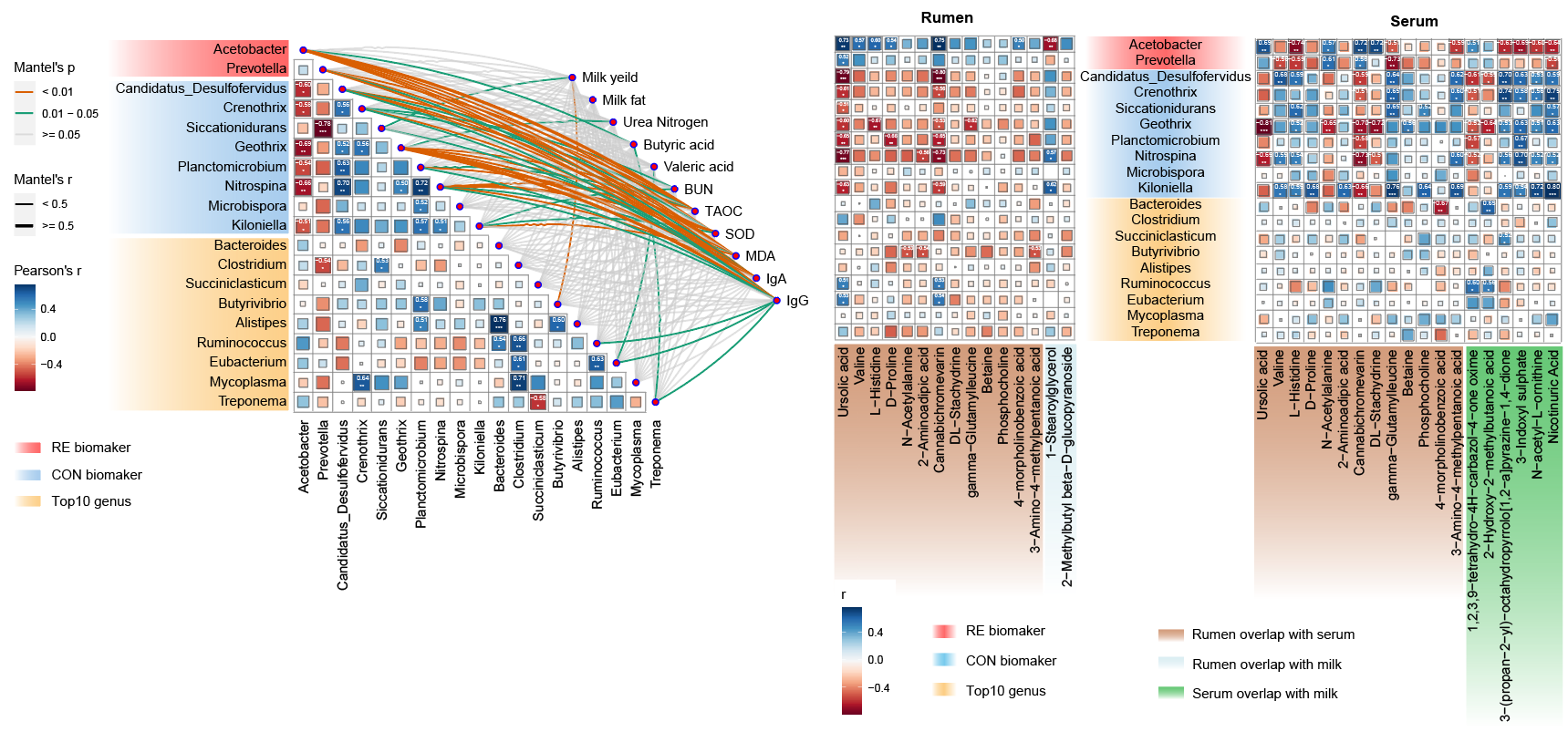
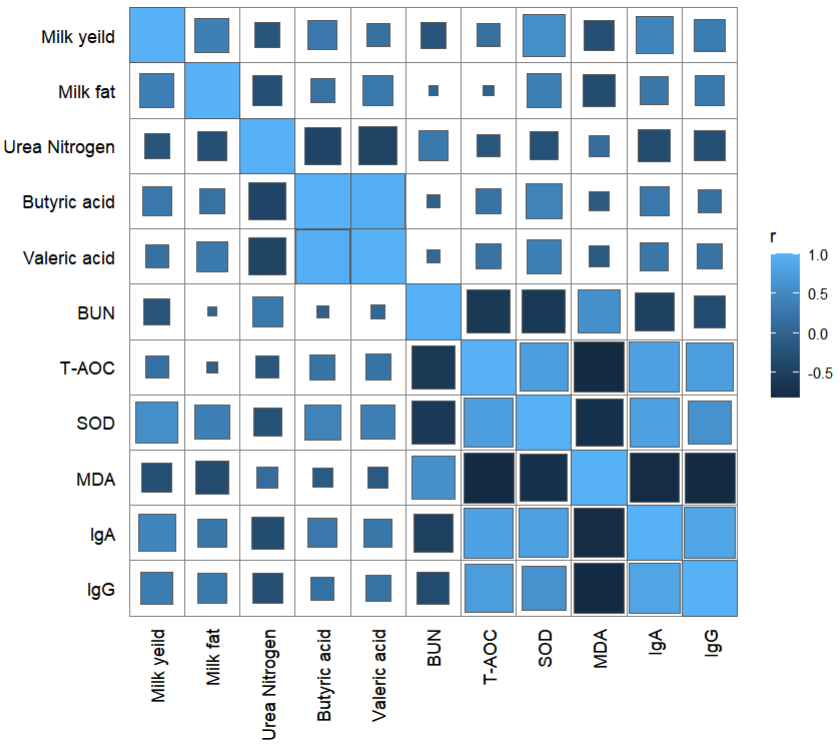
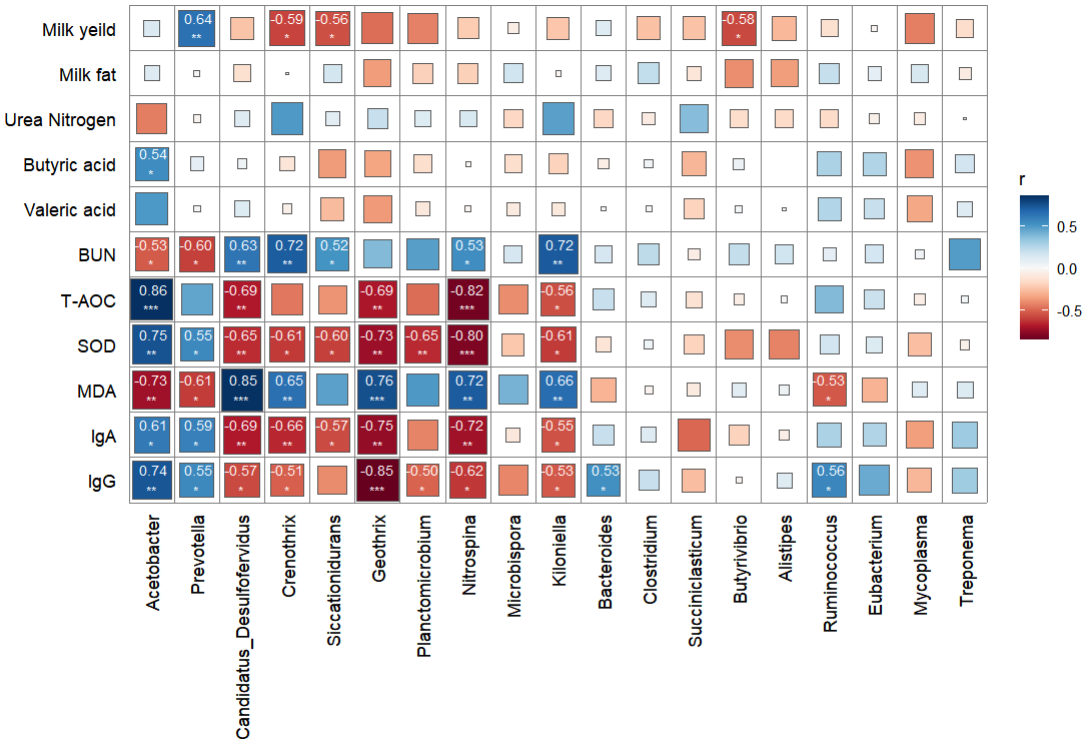
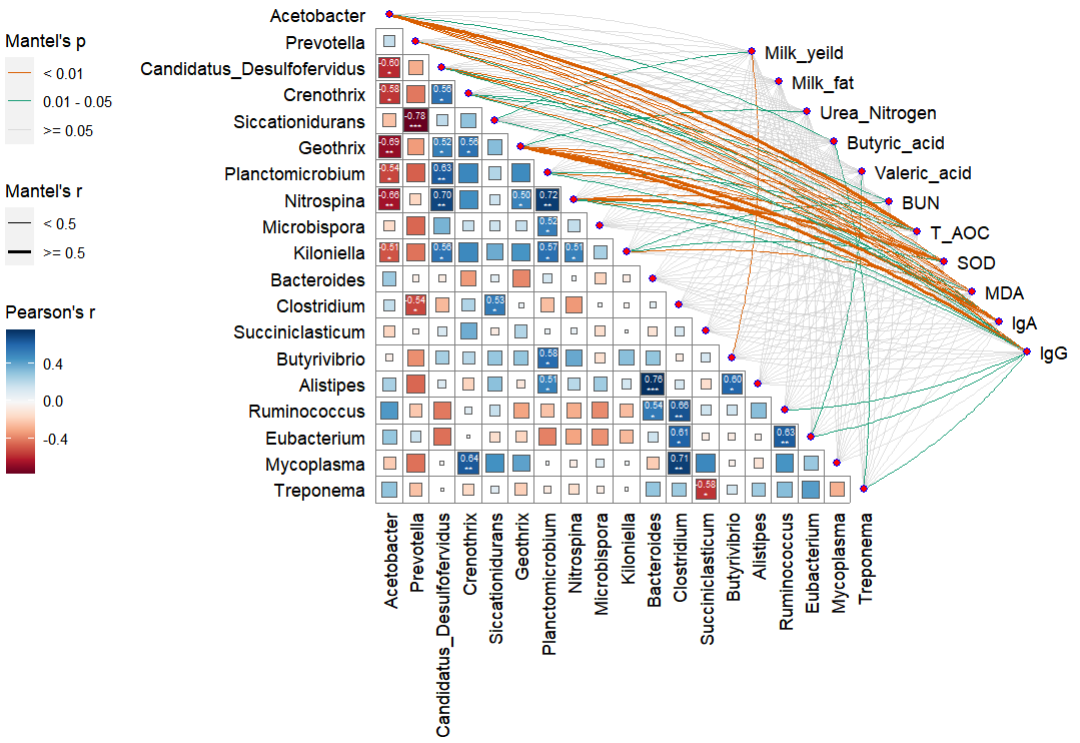
需求是对瘤胃宏基因组结果鉴定到的差异菌株与表观指标、瘤胃代谢组、血清代谢组、牛奶代谢组中有差异的部分进行关联分析,效果图如下:
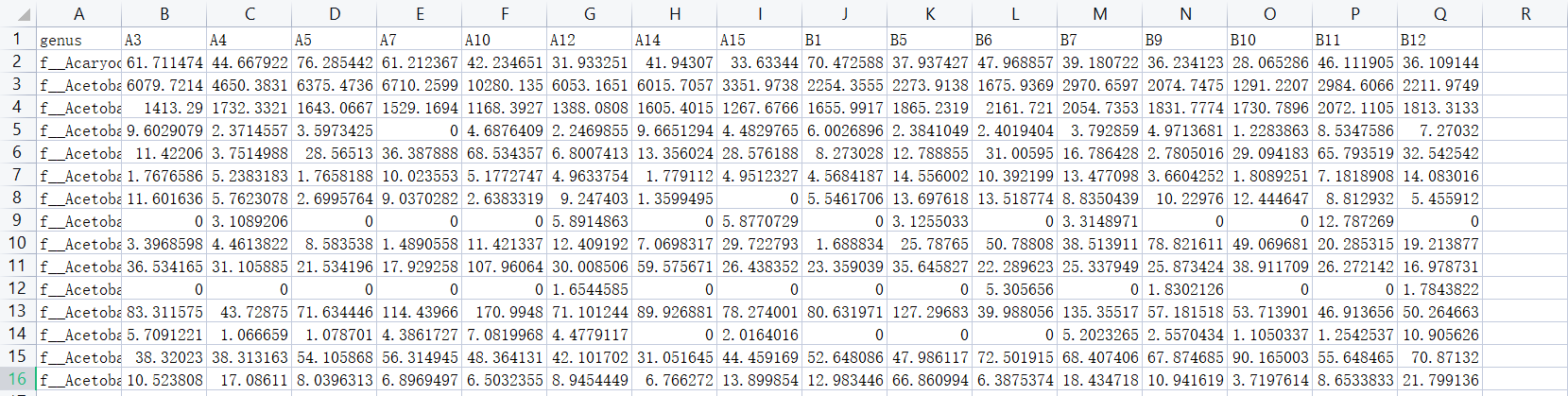
逗号分隔的csv格式文件,两个表格,一个是每个样本对应的表观指标数据,另一个是每个样本对应的菌群丰度,我这里用的是genus水平
rumen.csv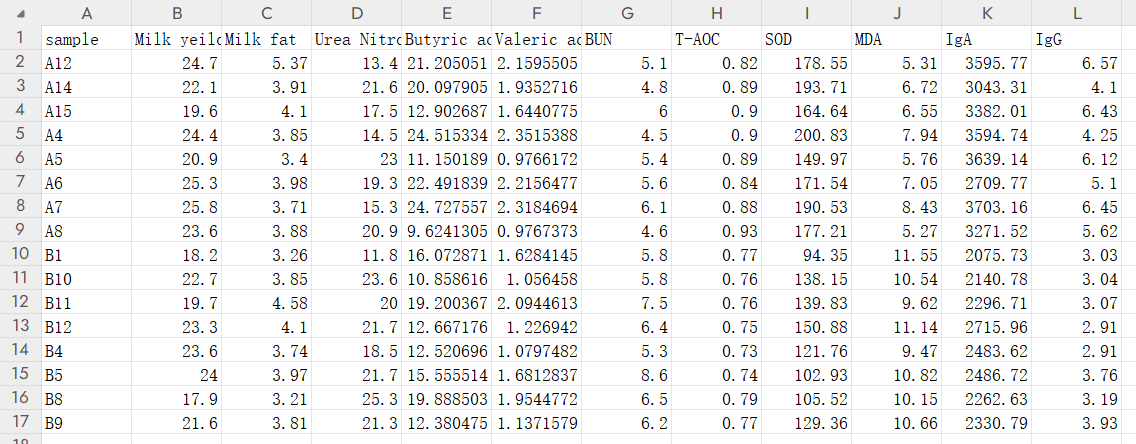
genus.csv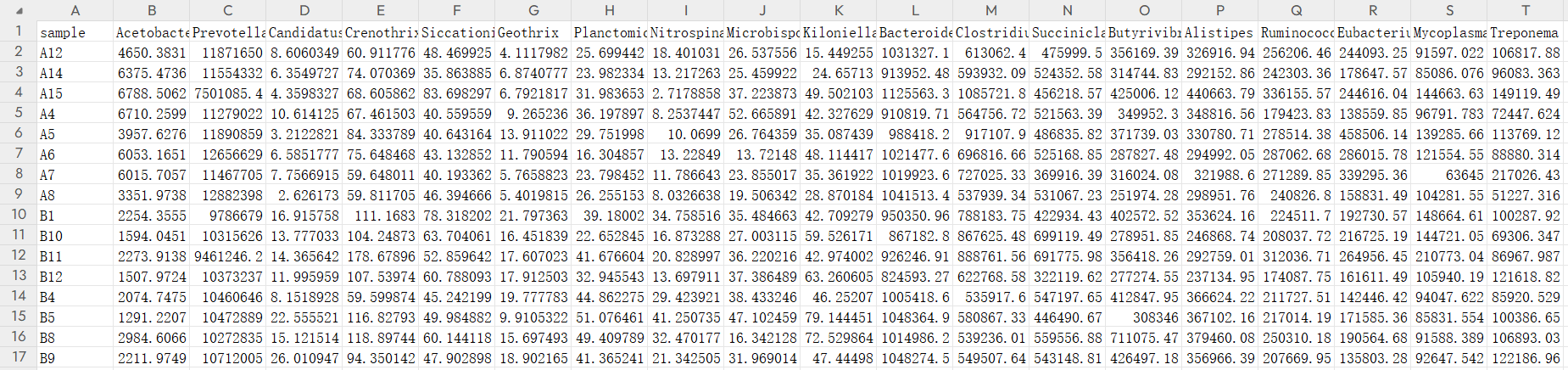
1 | install.pakages("linkET") |
如果报错R版本有问题装不上(我的4.3.1版本R出现了这个报错)请尝试:
1 | install.packages("devtools") |
1 | library(ggplot2) |
rumen.csv组内相关系数1 | matrix_data(list(rumen = rumen)) %>% |
1 | library(vegan) |
1 | library(dplyr) |
需求是返回的结果每组样本数量比较多,但组间差异不明显,挑出来重分析。详情请参考 [组间差异分析:Anosim]([组间差异分析:Anosim - 知乎 (zhihu.com)), 当然亲测更简便的方法可以借助云平台工具:

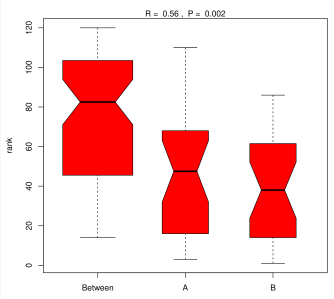
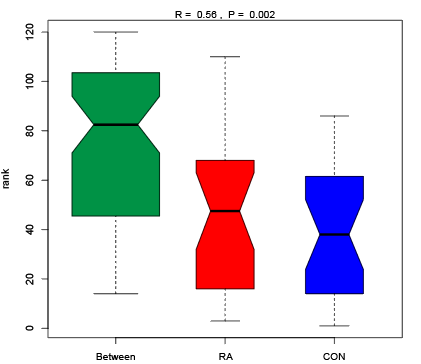

图形解读:盒型图纵轴表示距离排名,横轴between表示组间距离,其他表示对应分组组内距离。R值表示组间与组内的差异程度。R的范围为[-1,1]。R<0组间差异小于组内差异;R>0组间差异大于组内差异;R=0组间差异等于组内差异。
盒型图参数解释:
N:样本数;
Median:中位数
Q3:上四分位数
Q1:下四分位数
IOR:四分位距,interquartile range,即Q3-Q1
max:Q3+1.5IQR范围内的最大值
min:Q1-1.5IOR范围内的最小值
outlier:异常值/离群值,即小于Q1-1.5IOR或Q3+1.5IQR的值
notch:中位数95%置信区间(confidence intervals)的范围
同之前一样,重分析需要所以自己找了各路代码借鉴学习,详情请参考 R语言绘制带聚类树的堆叠柱形图 , 实操效果如下:
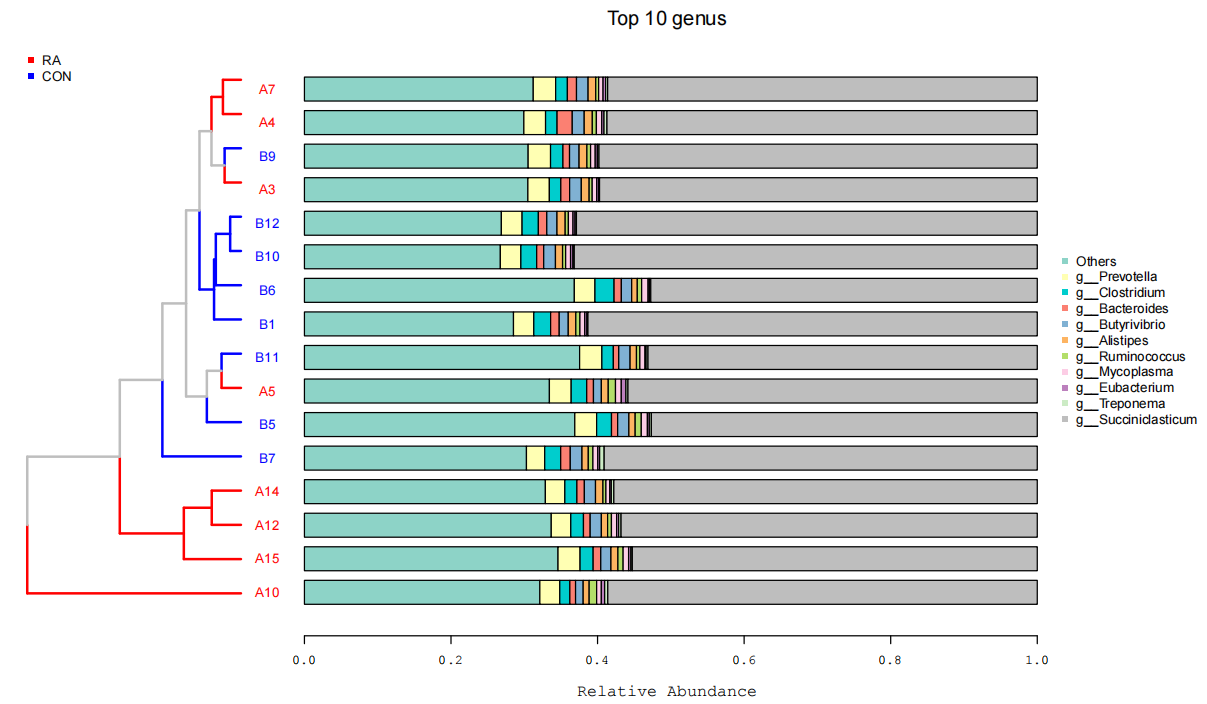
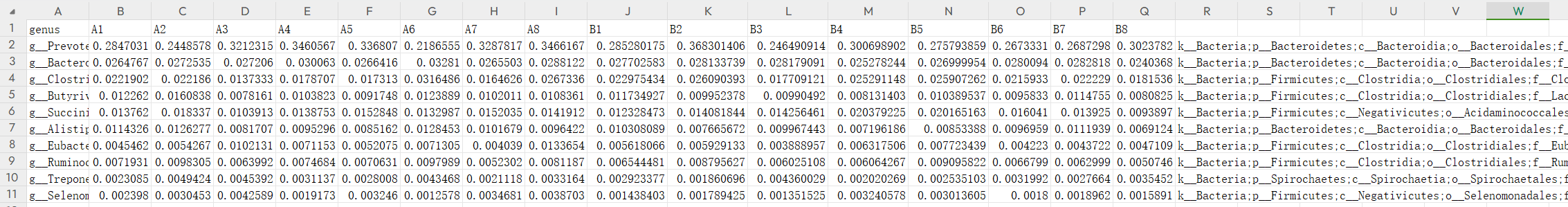
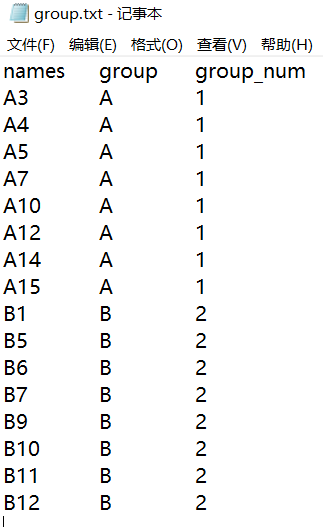
1 | # 装包 |
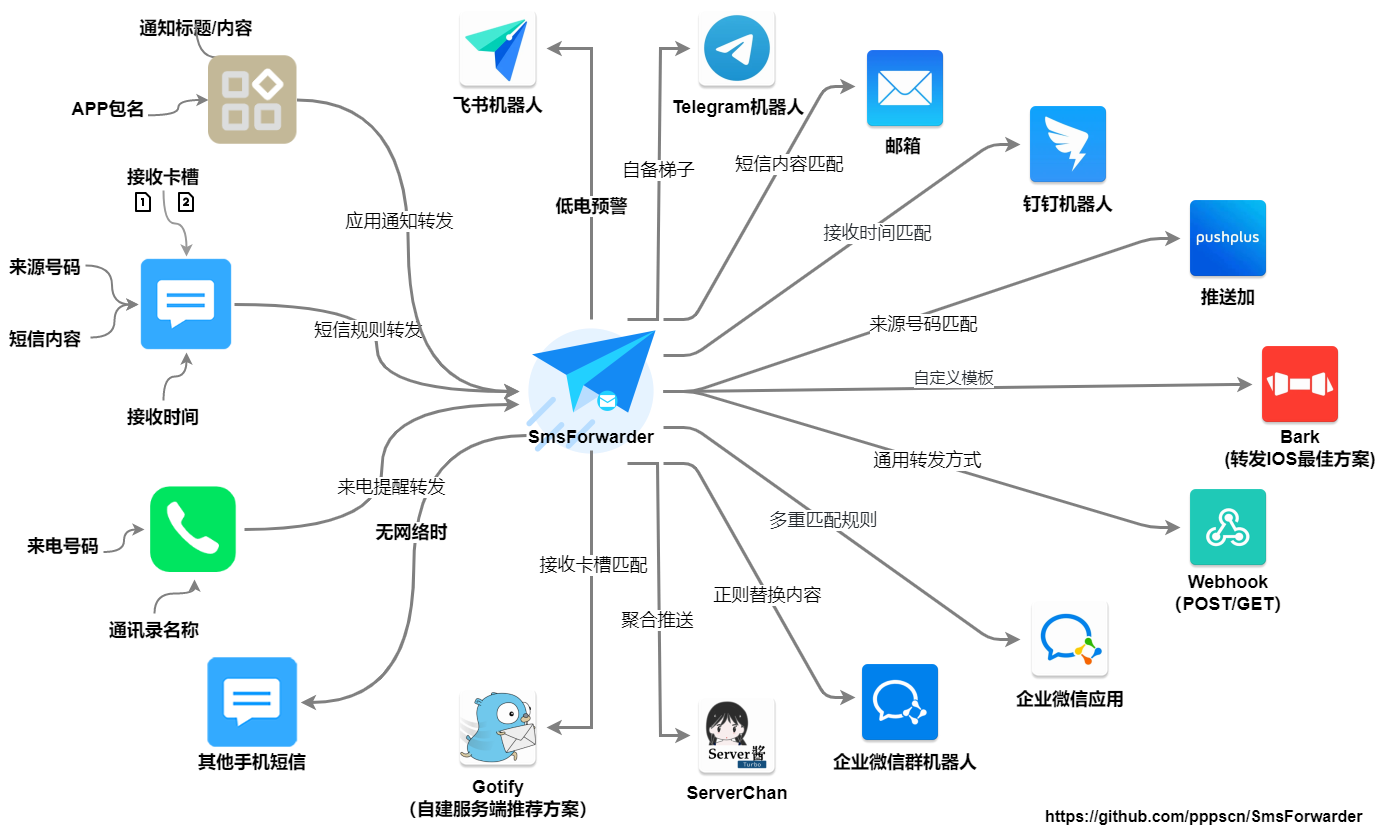
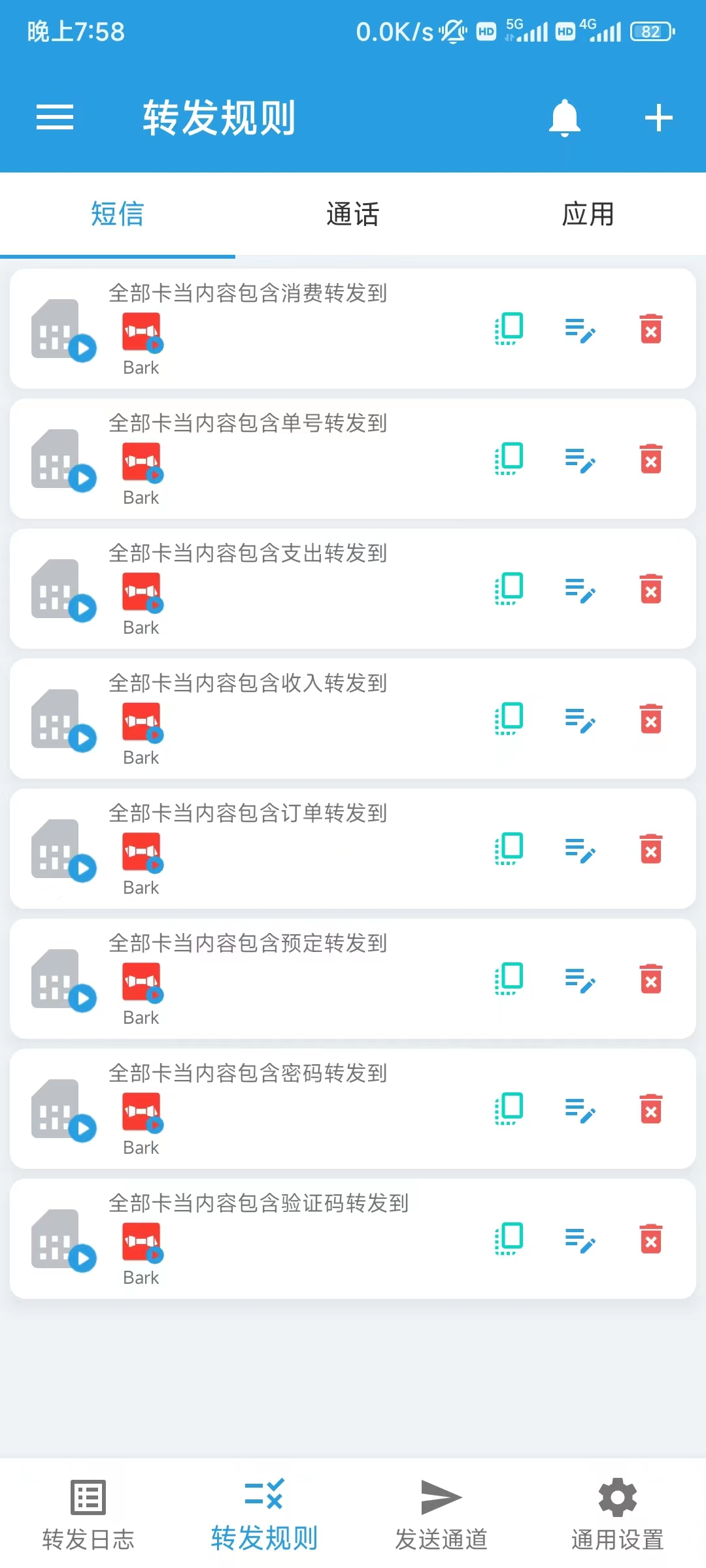
我的情况是现在手持两台设备,一台安卓(双卡),一台苹果(单卡),其中安卓机上的一个手机号开通保号套餐用于注册绑定账号以及接收短信,另外两张主要是流量卡功能。现在出于某些需要我有时只带苹果机出门,现在有个需求是需要用安卓机接收短信,然后安卓机不在身边的时候也可以及时转发到苹果机上。
这里就可以用到一个APP:SmsForwarder-短信转发器

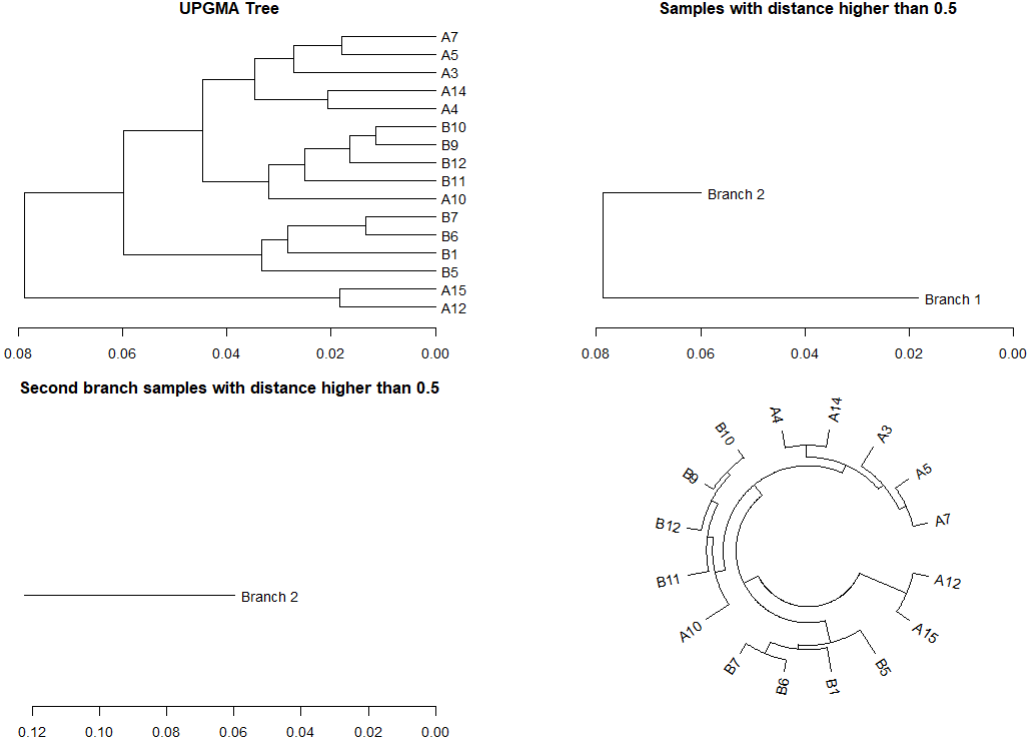
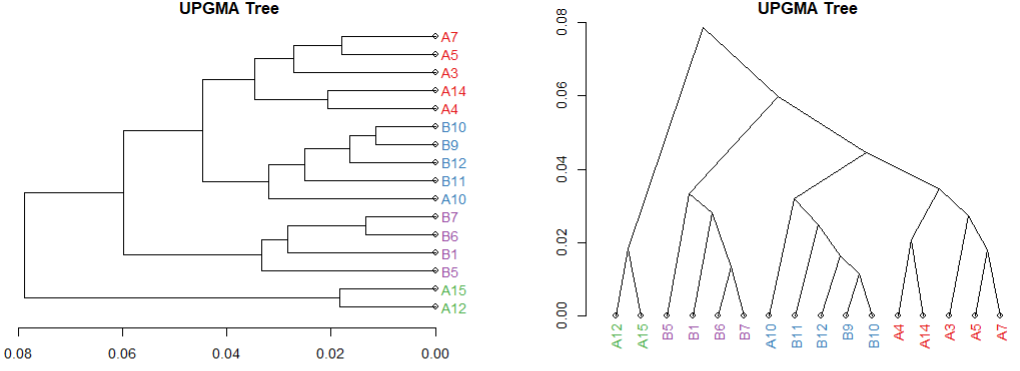
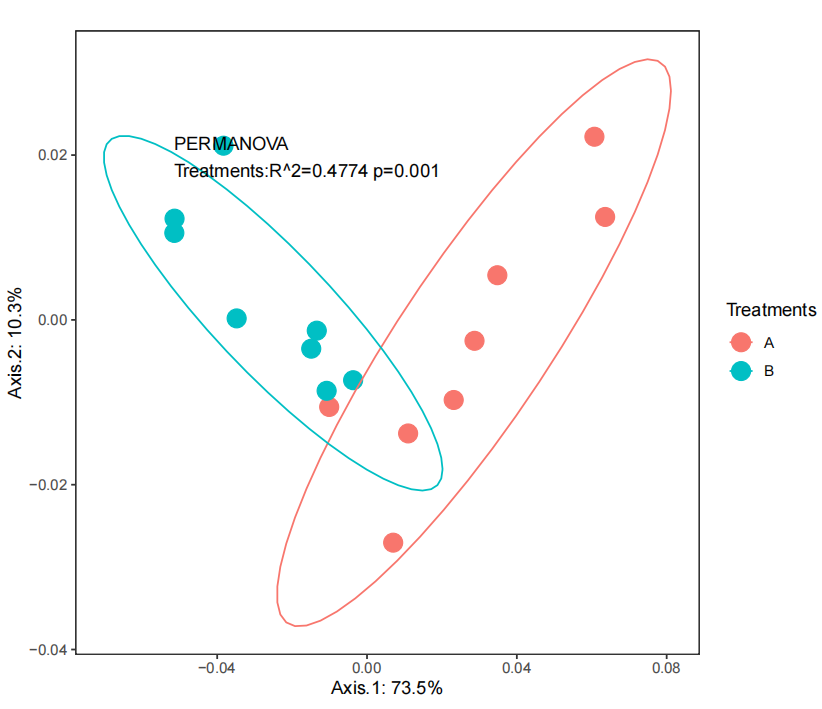
同上一篇的PCoA分析,这个也是基于公司结果基础上的再次分析,重新挑选样本,在公司结果提供的csv结果表上进行删减,本地重新分析作图
1 | install.packages('vegan') |
1 |
|
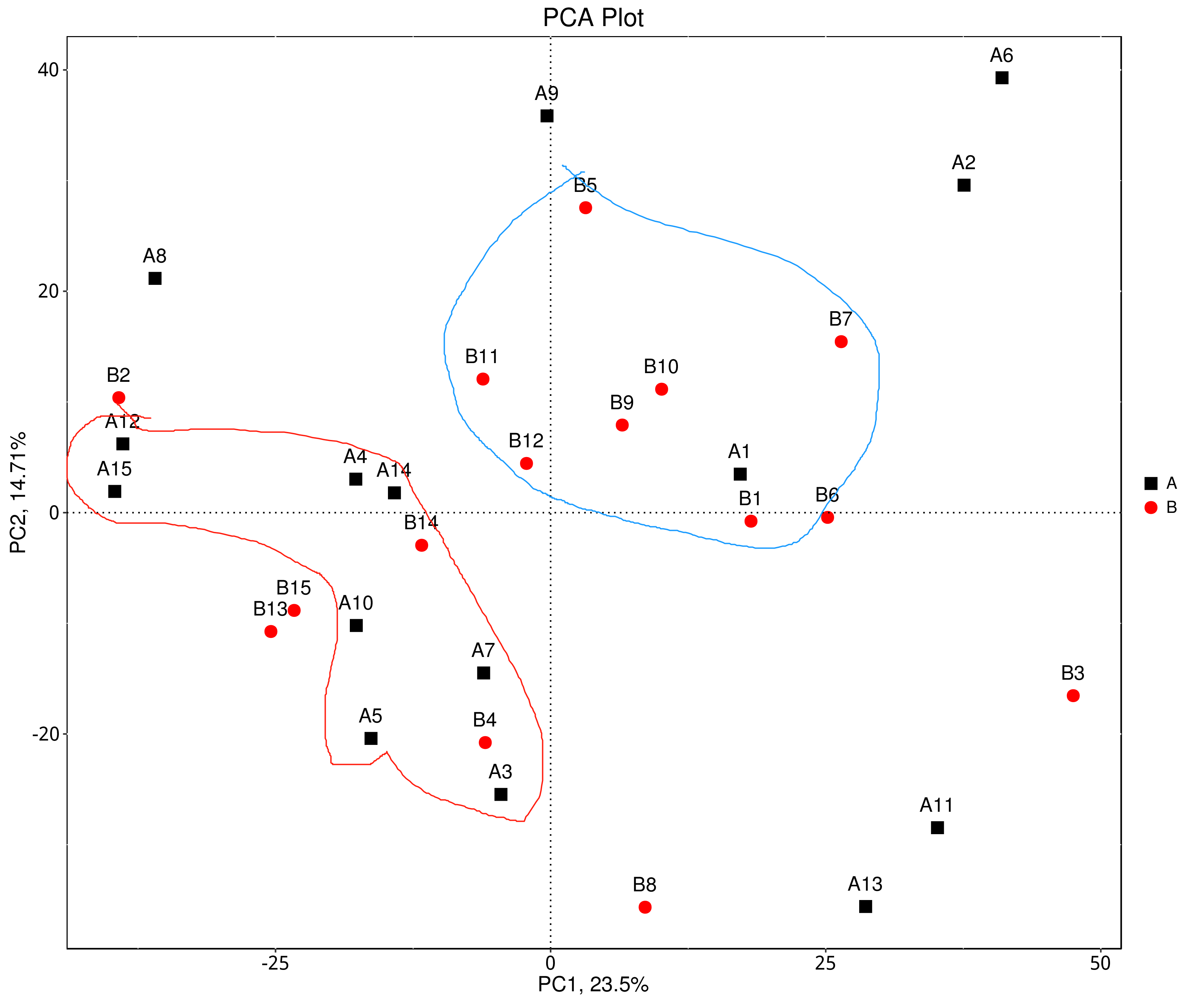
因为公司给的PCA结果效果不佳,决定从中重新挑选部分样本进行再分析
1 | library(vegan) # 计算bray距离 |
缺失模块。
1、请确保node版本大于6.2
2、在博客根目录(注意不是yilia根目录)执行以下命令:
npm i hexo-generator-json-content --save
3、在根目录_config.yml里添加配置:
jsonContent:
meta: false
pages: false
posts:
title: true
date: true
path: true
text: false
raw: false
content: false
slug: false
updated: false
comments: false
link: false
permalink: false
excerpt: false
categories: false
tags: true
