写在前面
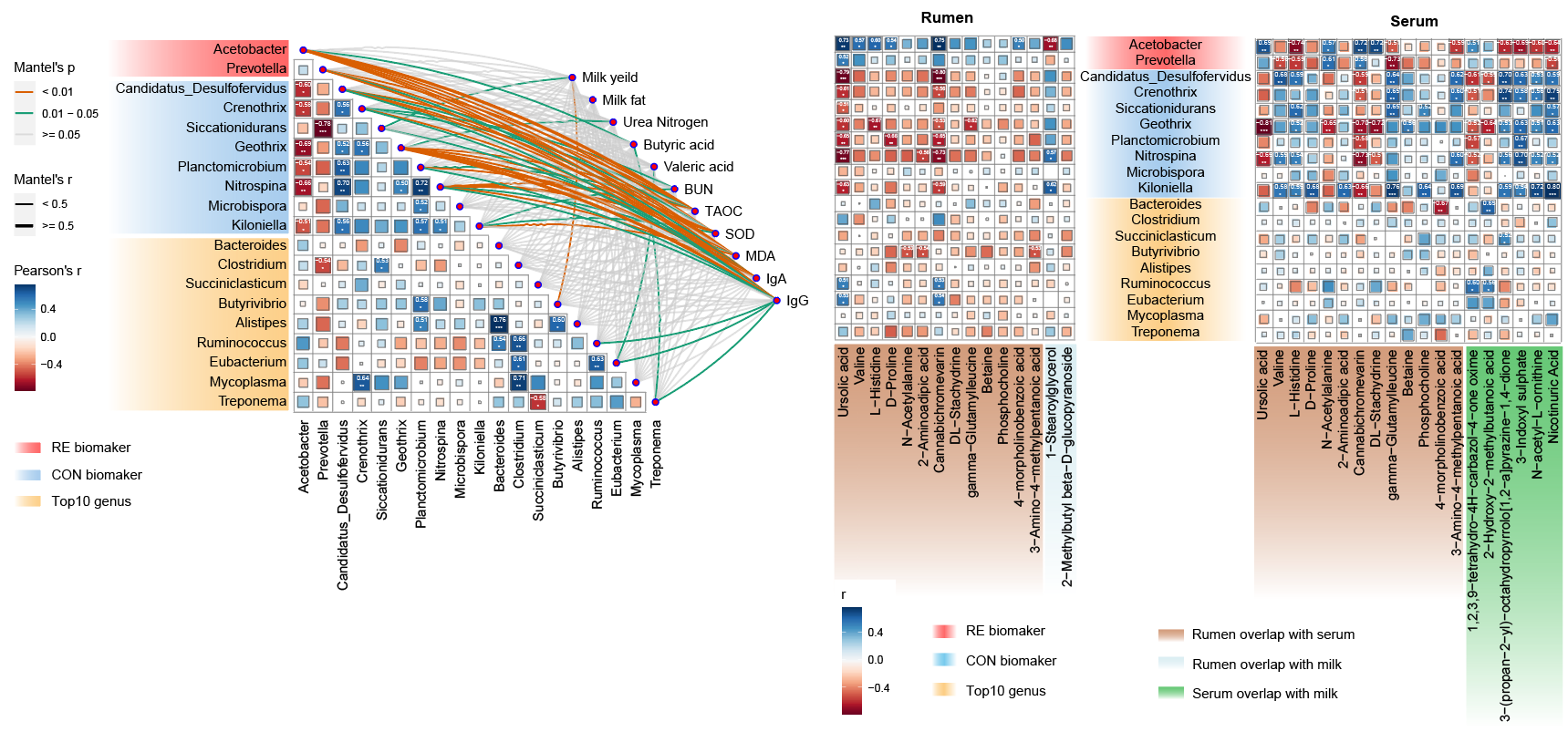
需求是对瘤胃宏基因组结果鉴定到的差异菌株与表观指标、瘤胃代谢组、血清代谢组、牛奶代谢组中有差异的部分进行关联分析,效果图如下:
数据准备
逗号分隔的csv格式文件,两个表格,一个是每个样本对应的表观指标数据,另一个是每个样本对应的菌群丰度,我这里用的是genus水平
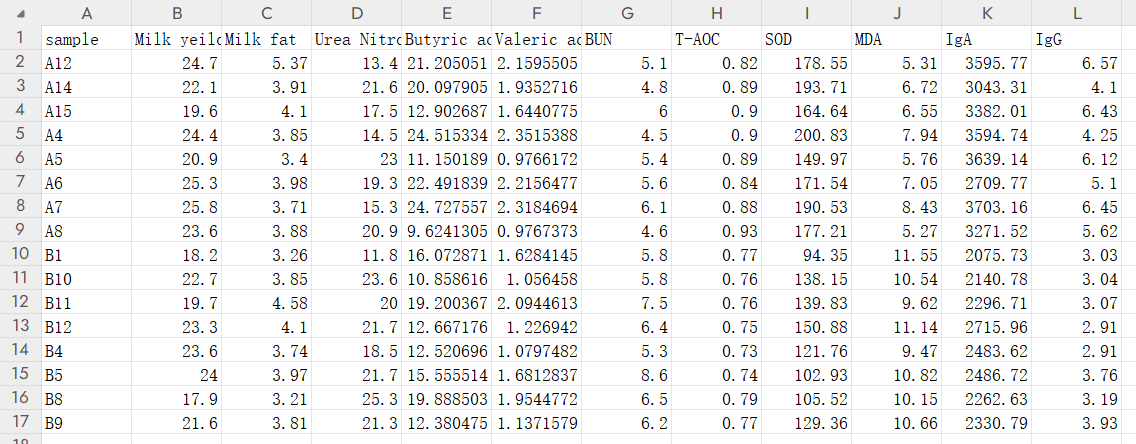
- 需要关联的表观数据
rumen.csv
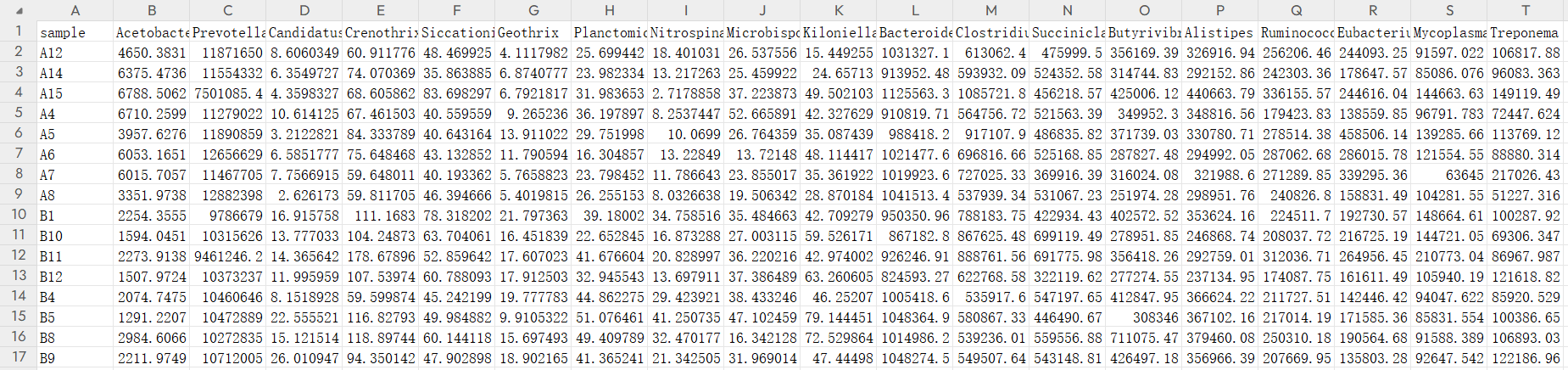
- 不同样本的菌群丰度
genus.csv
R包linkET可视化
- 装包
1 | install.pakages("linkET") |
如果报错R版本有问题装不上(我的4.3.1版本R出现了这个报错)请尝试:
1 | install.packages("devtools") |
- 读取数据
1 | library(ggplot2) |
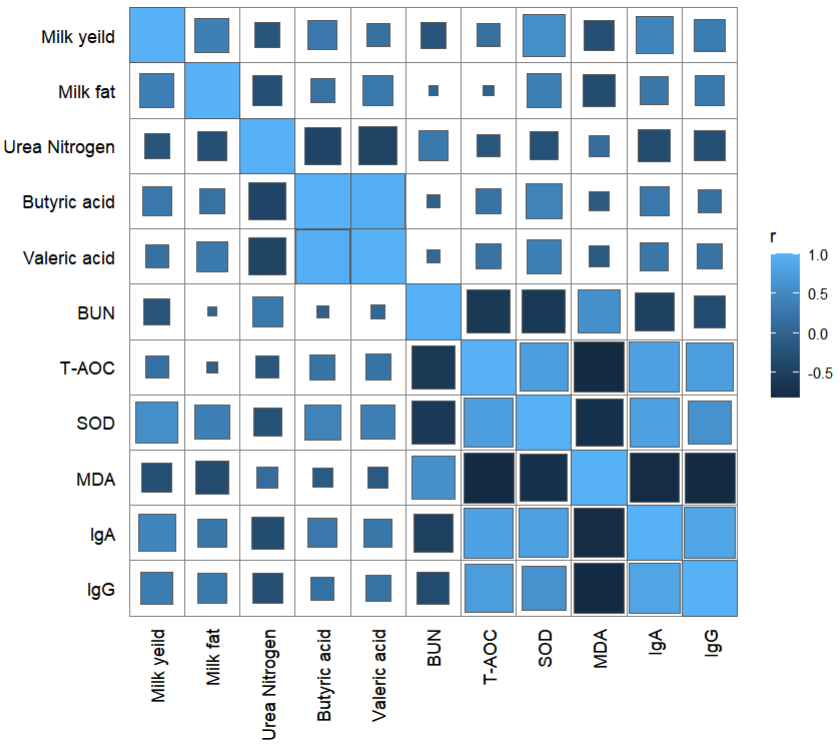
rumen.csv组内相关系数
1 | matrix_data(list(rumen = rumen)) %>% |
- 两个表格进行关联生成相关性矩阵图,带显著性标记
1 | library(vegan) |
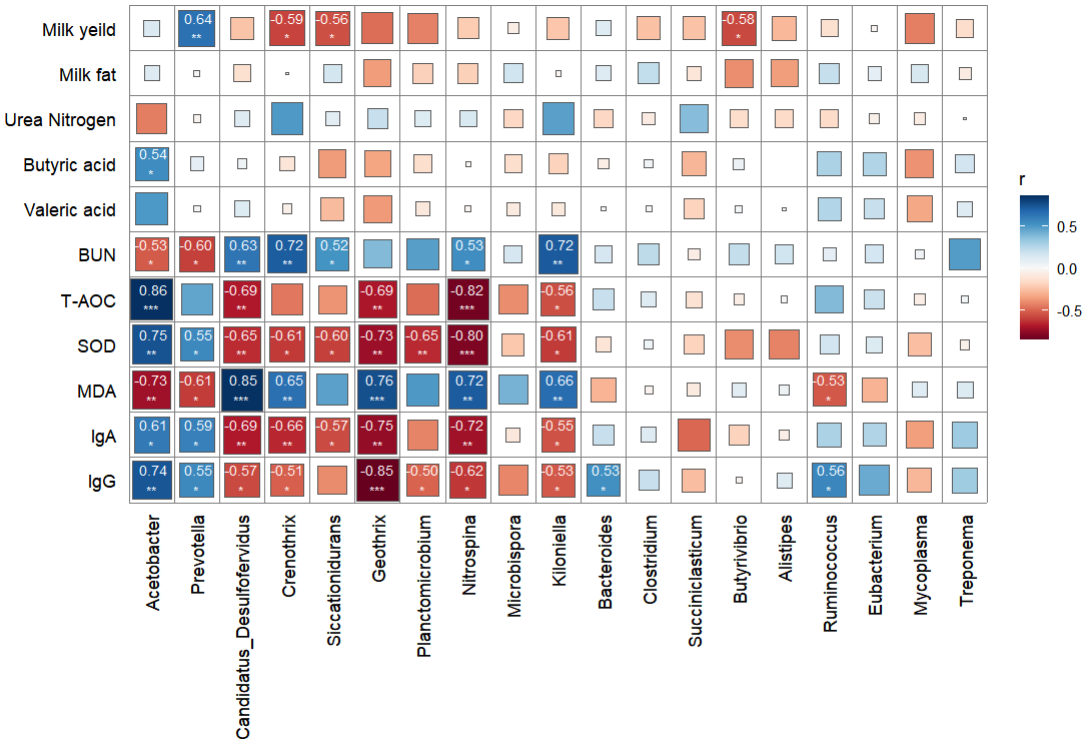
- 加工可视化
1 | library(dplyr) |
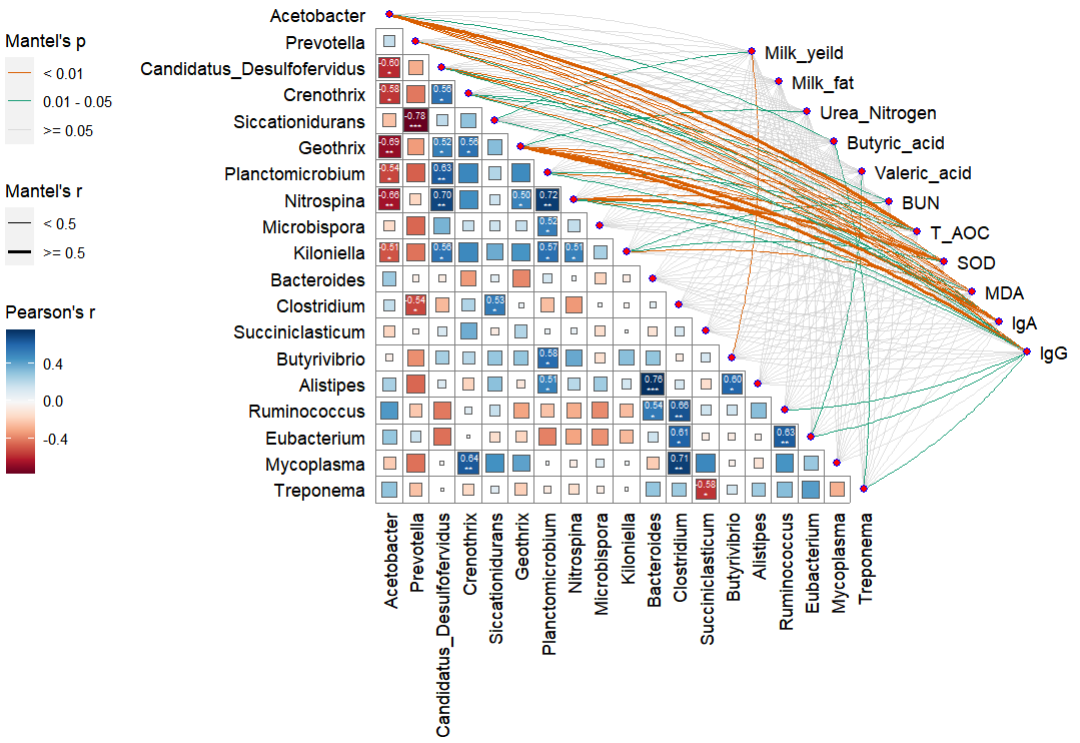
- 不显著的灰色连接线部分也可以去掉让画面更干净。其余细节去AI加工即可。
本文作者:
twocanis
本文链接: https://twocanis.github.io/2023/09/26/%E5%9F%BA%E4%BA%8ER%E7%9A%84linkET%E5%8C%85%E5%8F%AF%E8%A7%86%E5%8C%96%E7%9B%B8%E5%85%B3%E6%80%A7%E7%BD%91%E7%BB%9C%E7%83%AD%E5%9B%BE%E5%88%86%E6%9E%90correlation-heatmap/
版权声明: 本作品采用 知识共享署名-非商业性使用-相同方式共享 4.0 国际许可协议 进行许可。转载请注明出处!

本文链接: https://twocanis.github.io/2023/09/26/%E5%9F%BA%E4%BA%8ER%E7%9A%84linkET%E5%8C%85%E5%8F%AF%E8%A7%86%E5%8C%96%E7%9B%B8%E5%85%B3%E6%80%A7%E7%BD%91%E7%BB%9C%E7%83%AD%E5%9B%BE%E5%88%86%E6%9E%90correlation-heatmap/
版权声明: 本作品采用 知识共享署名-非商业性使用-相同方式共享 4.0 国际许可协议 进行许可。转载请注明出处!



