写在前面
拿到多组学的数据后一直在找合适的方法将二者进行关联,比如我这里是三种体液的代谢组与一种体液的宏基因组。需求是对多组学进行关联分析,直到最近看到不少文章里利用Gephi将相关性表格进行可视化的图,效果还不错,于是写个过程记录自用。这里演示的是属水平的差异菌群相对丰度(宏基因组结果)与代谢组鉴定到的差异代谢物进行关联。
主要分为两个部分:
- 先是计算相关性生成
graphml格式文件 - 使用Gephi软件进行可视化
数据准备
主要就是两个需要关联的表格
差异代谢物表格
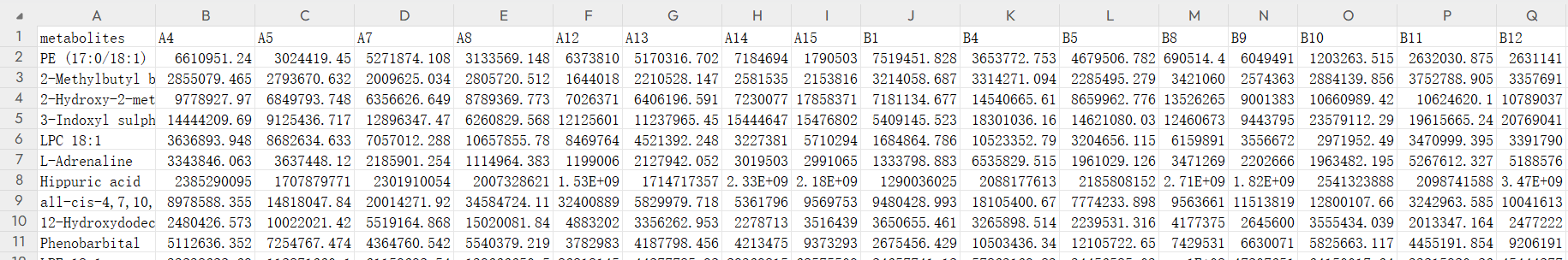
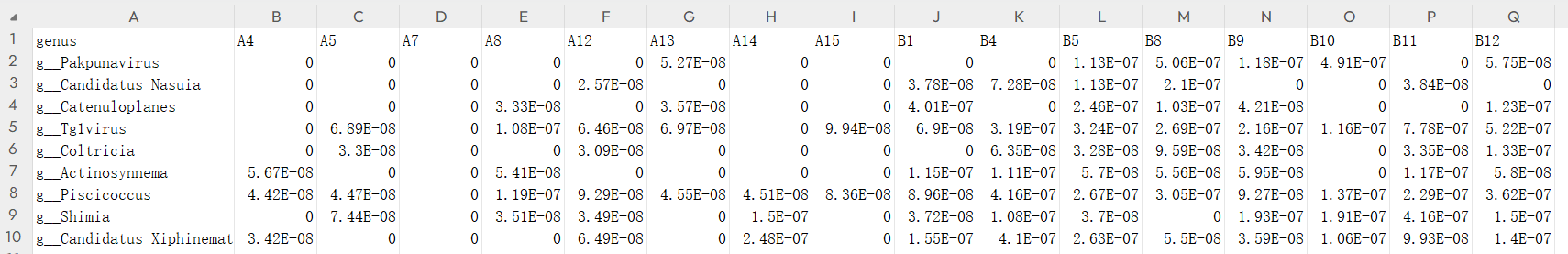
差异菌属的相对丰度表格
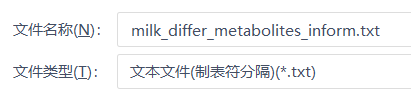
这里注意:原文件如果是新建的excel,默认有3个sheet,一定记得只保留一个sheet后另存为制表符分隔的txt文件,这一点很重要,不然下面分析的时候会从读取就开始报错!
R包Hmisc计算相关性并用igraph包生成graphml
用到Hmisc包和igraph包
这里就直接放代码吧,遇到相关的报错先仔细检查数据,一般是数据格式和内容本身的问题。不行就上网找解决办法。
1 | ###微生物和代谢物的相关性网络 |
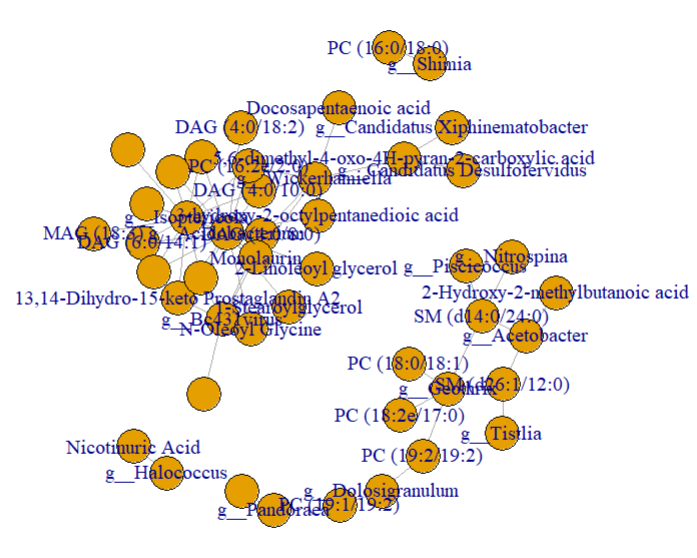
plot包出的图
Gephi软件可视化
下载地址:The Open Graph Viz Platform,支持中文
可参考 B站:Gephi可视化进阶:教你绘制一个漂亮的网络图
进一步对上面生成的network.graphml文件用Gephi可视化
- 导入图形文件
- 导入后初始布局
- 更改布局,等节点的位置没有移动变化即可停止
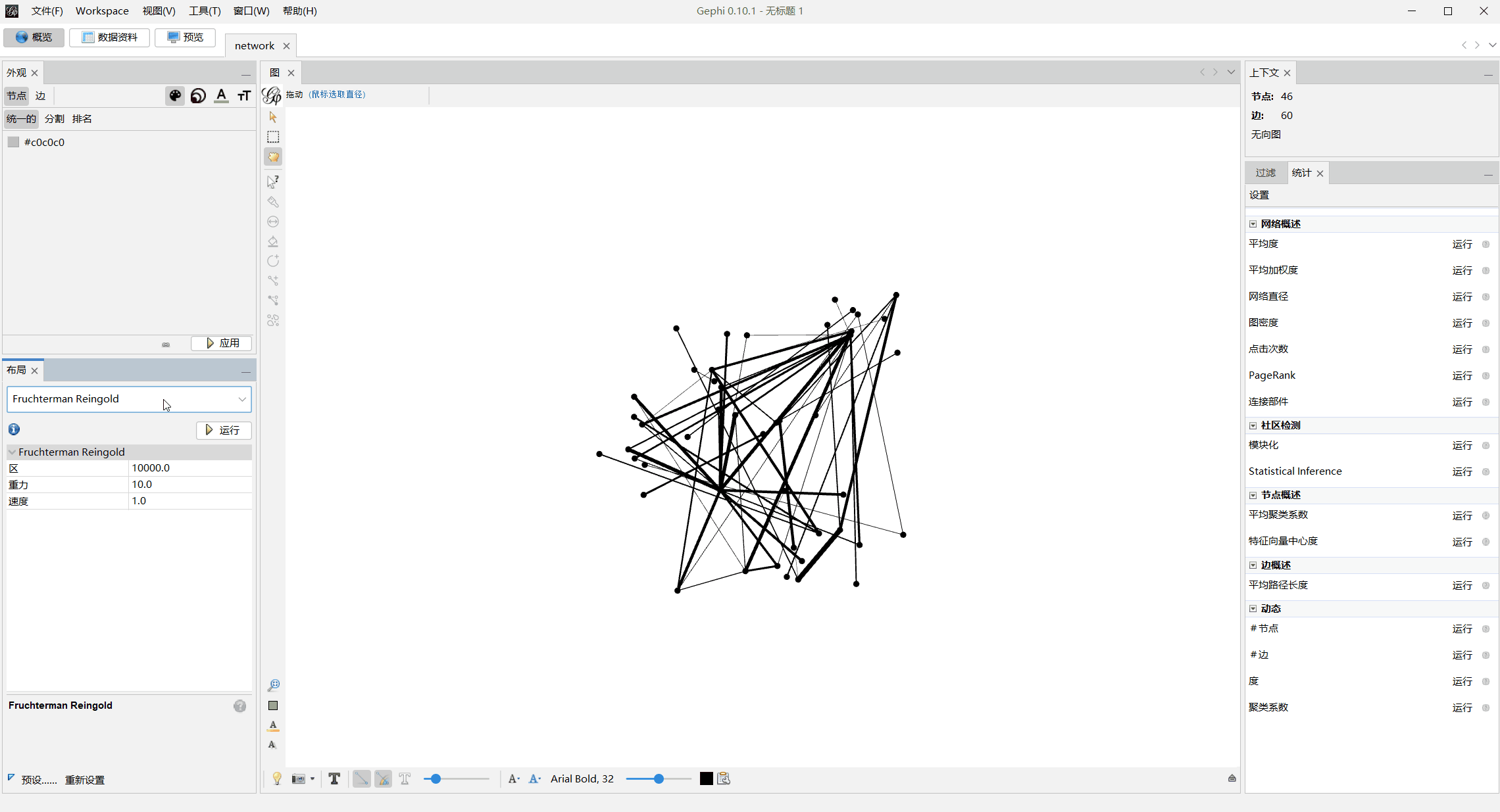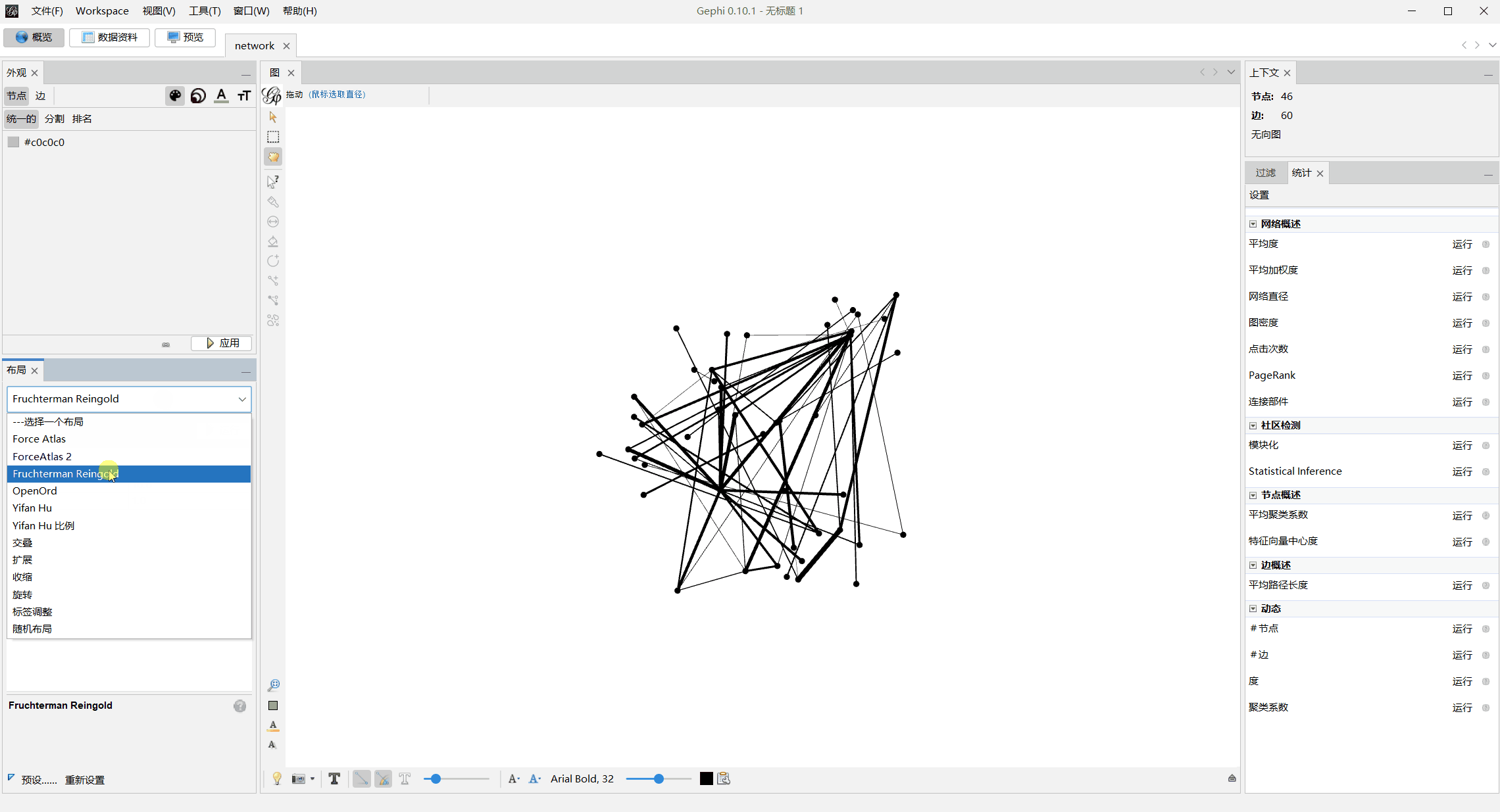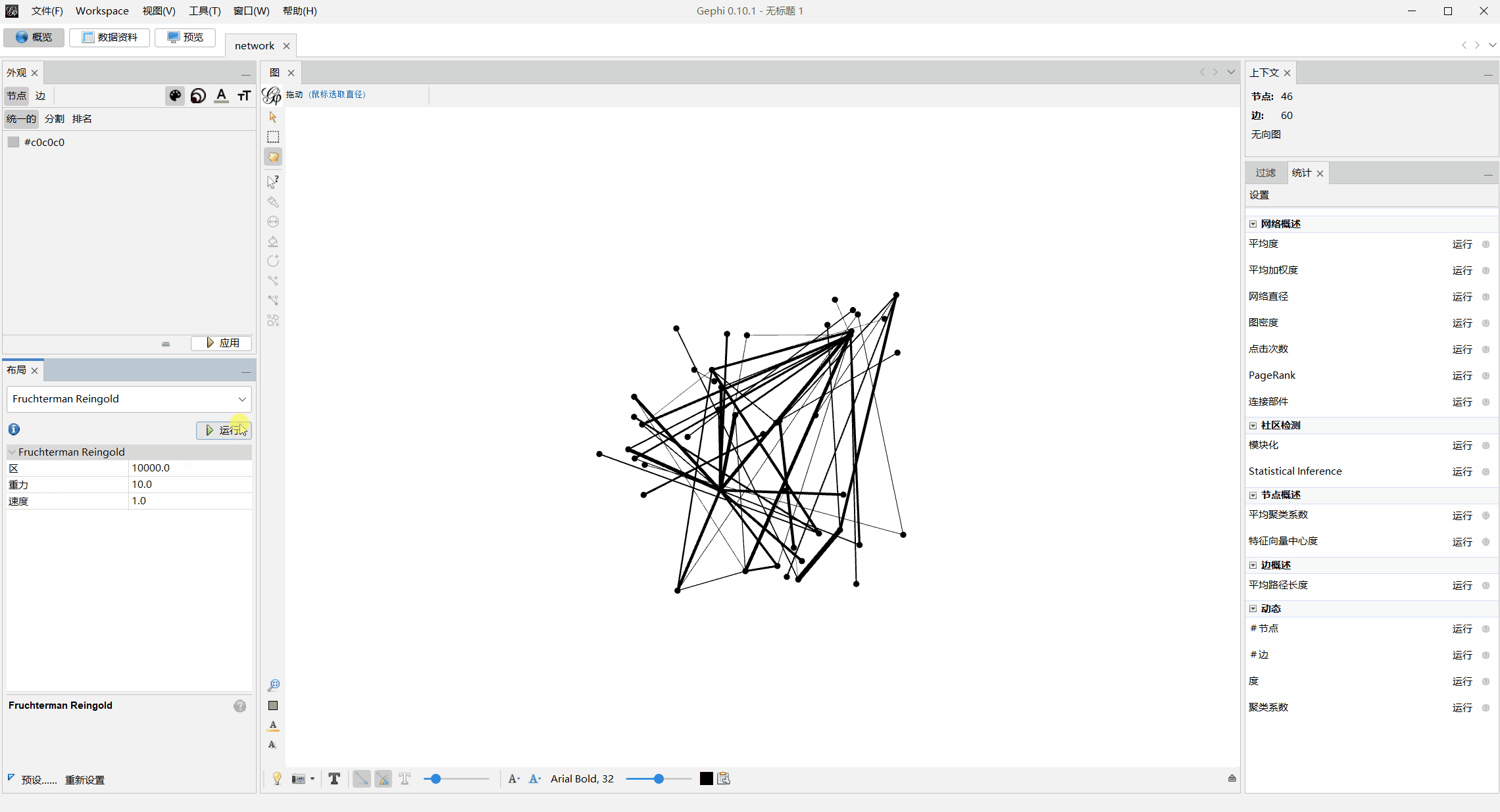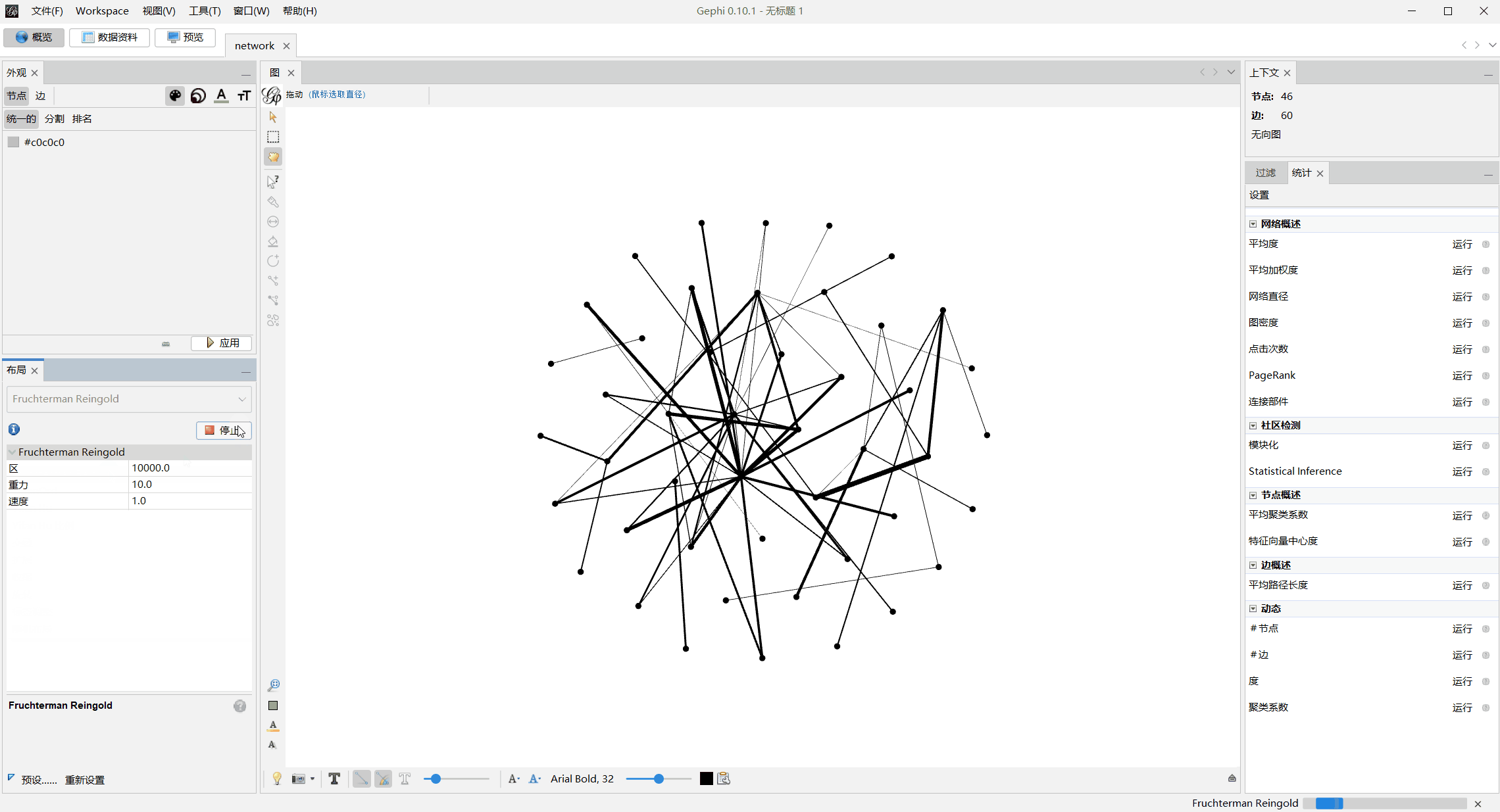
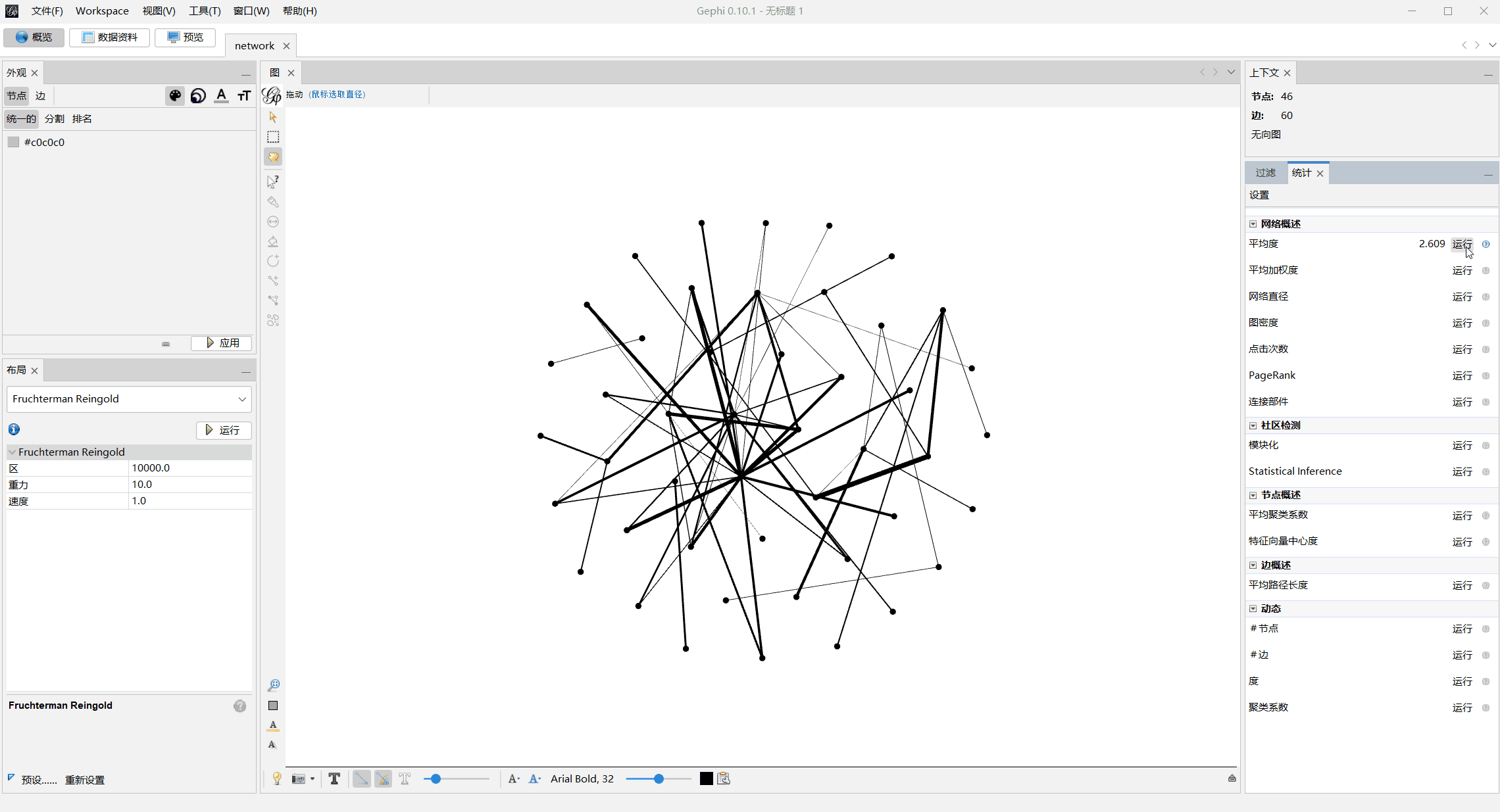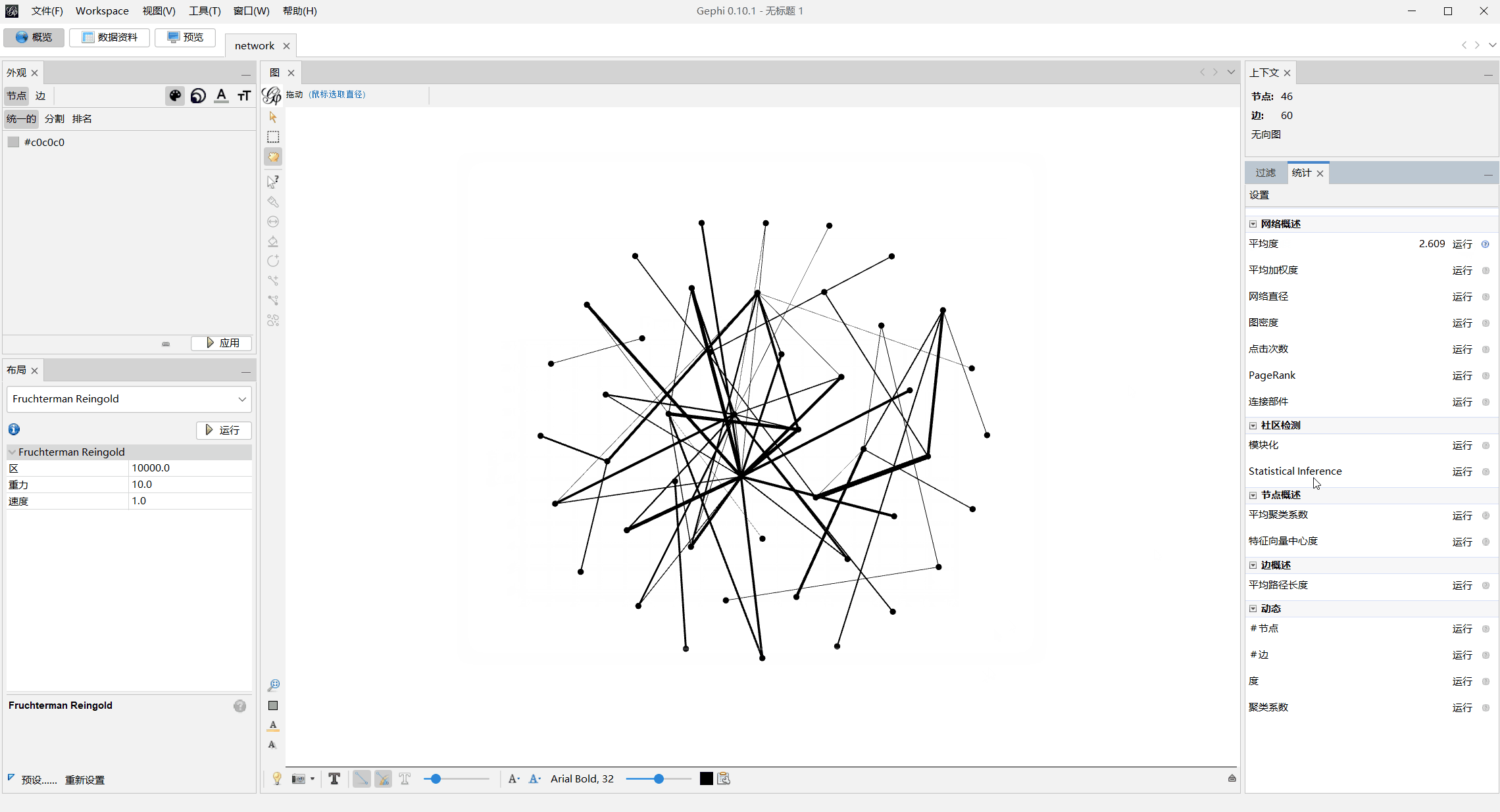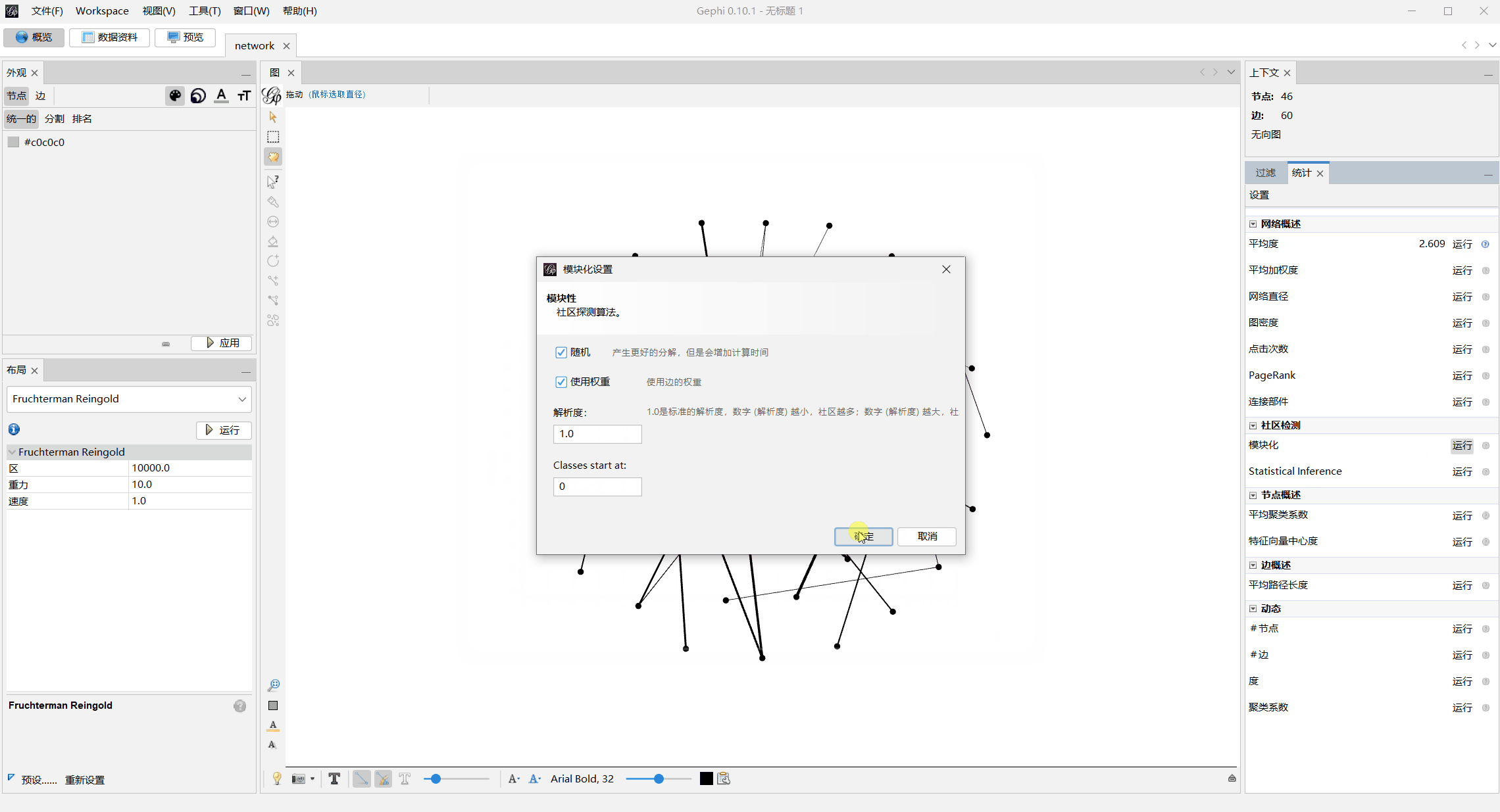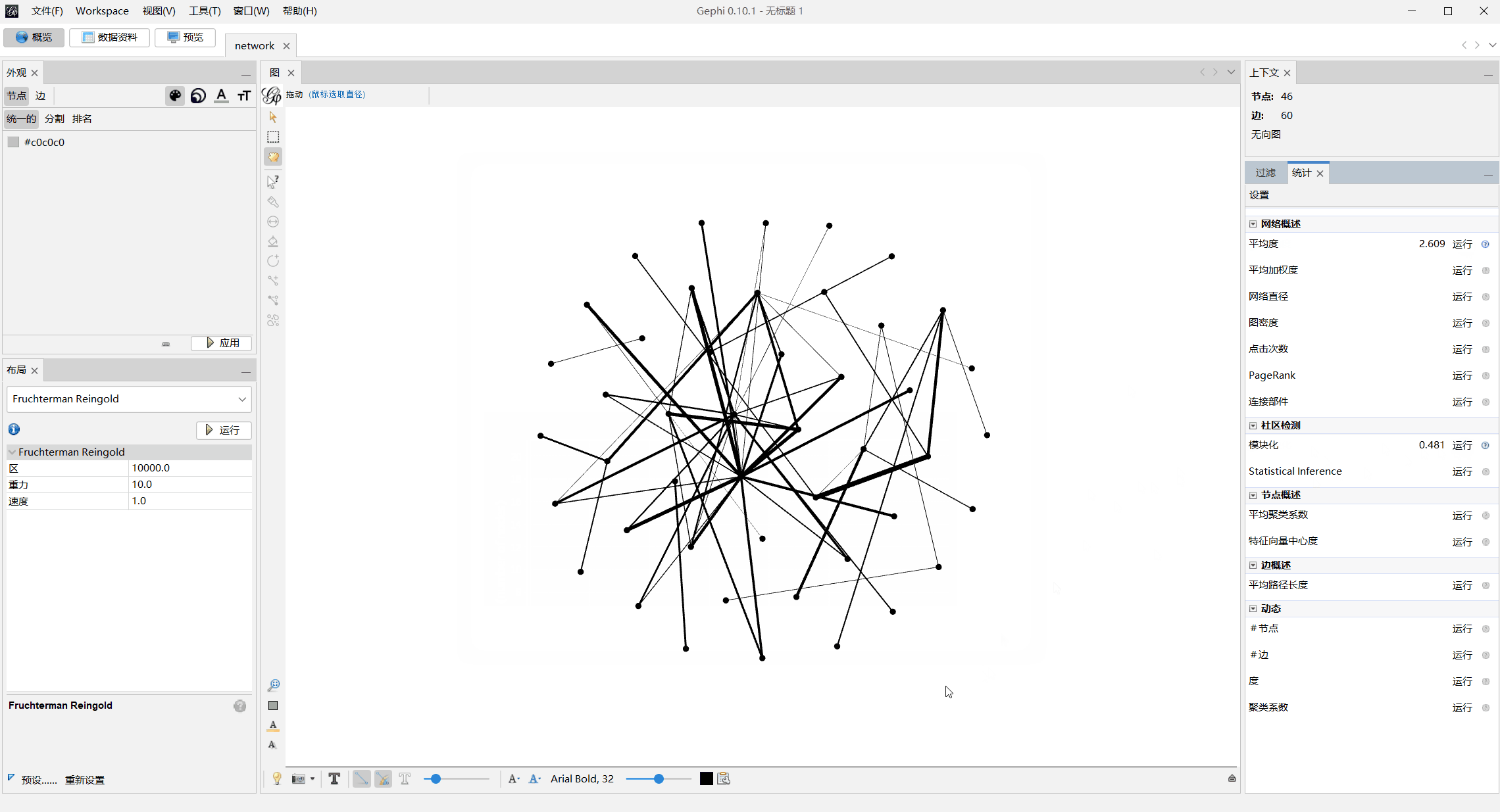
- 计算平均度与模块化
模块化上色
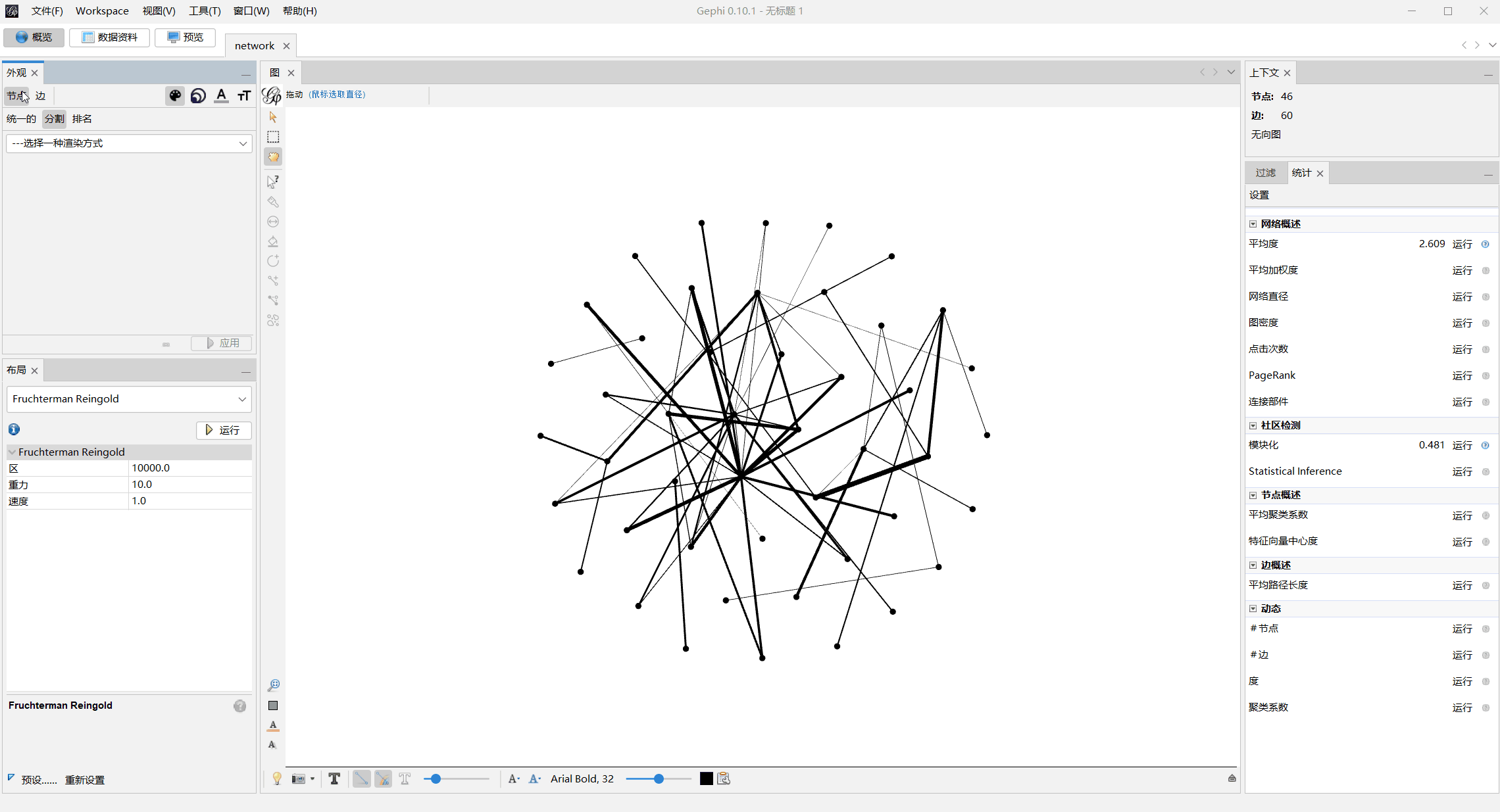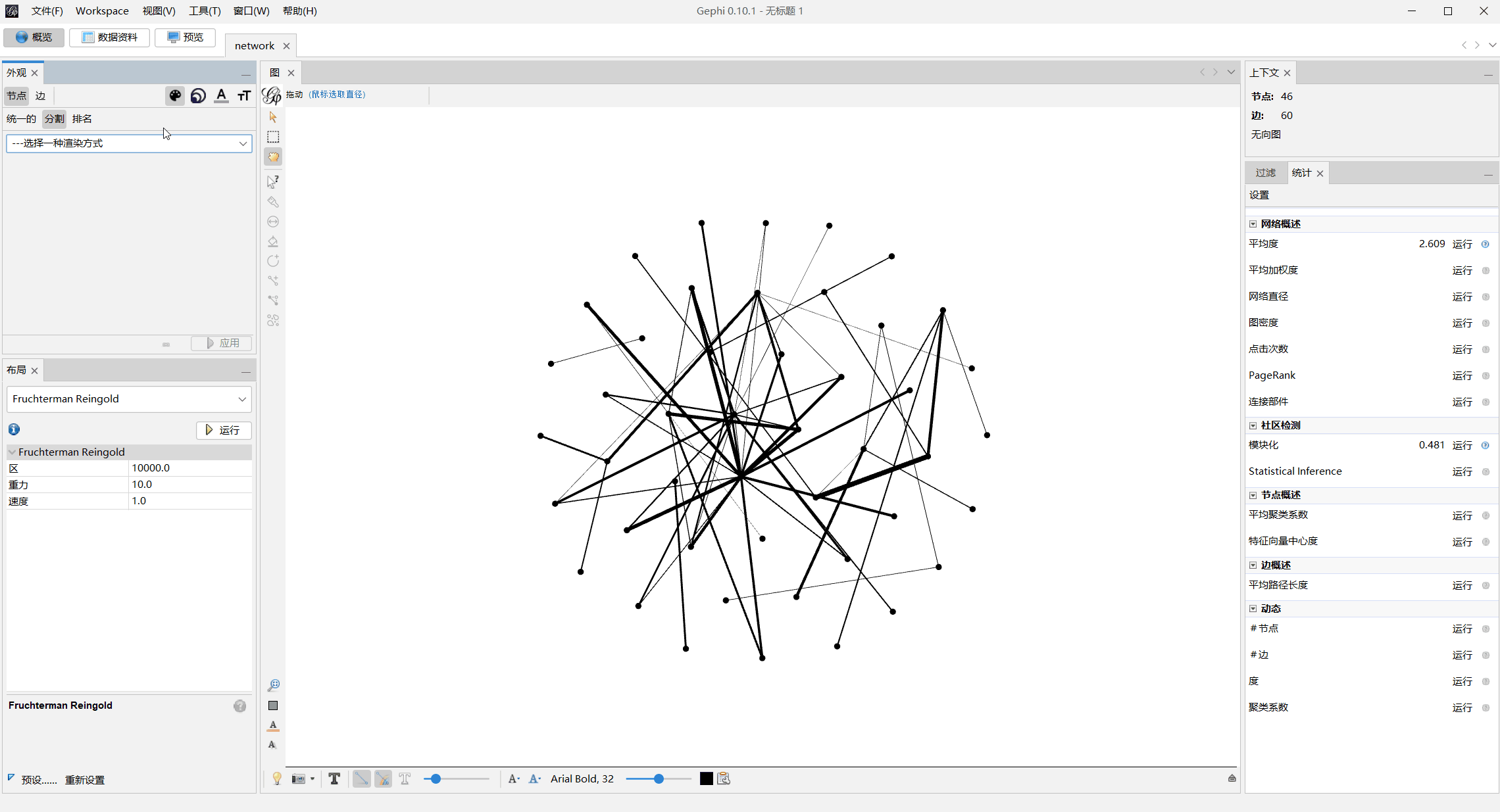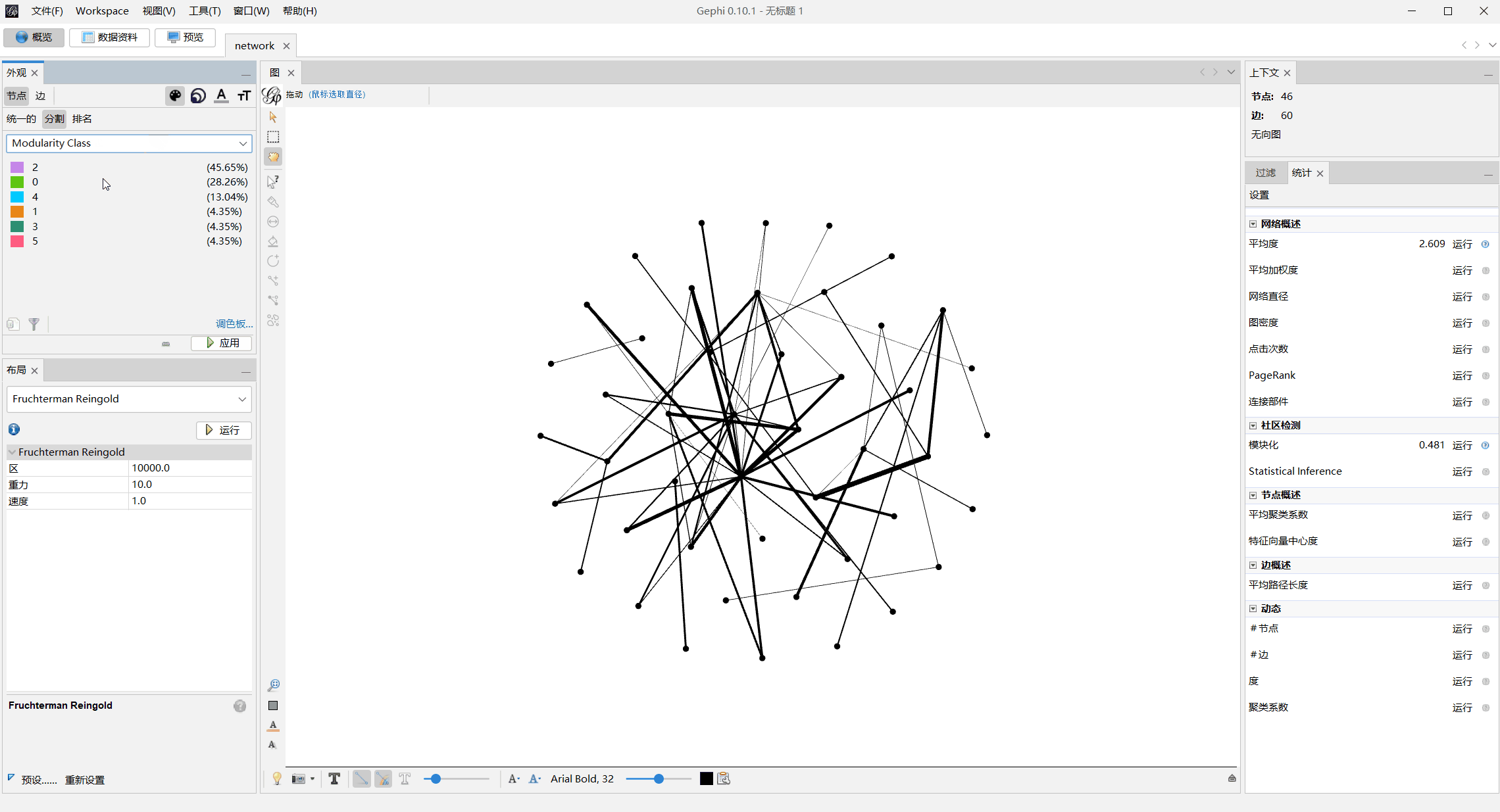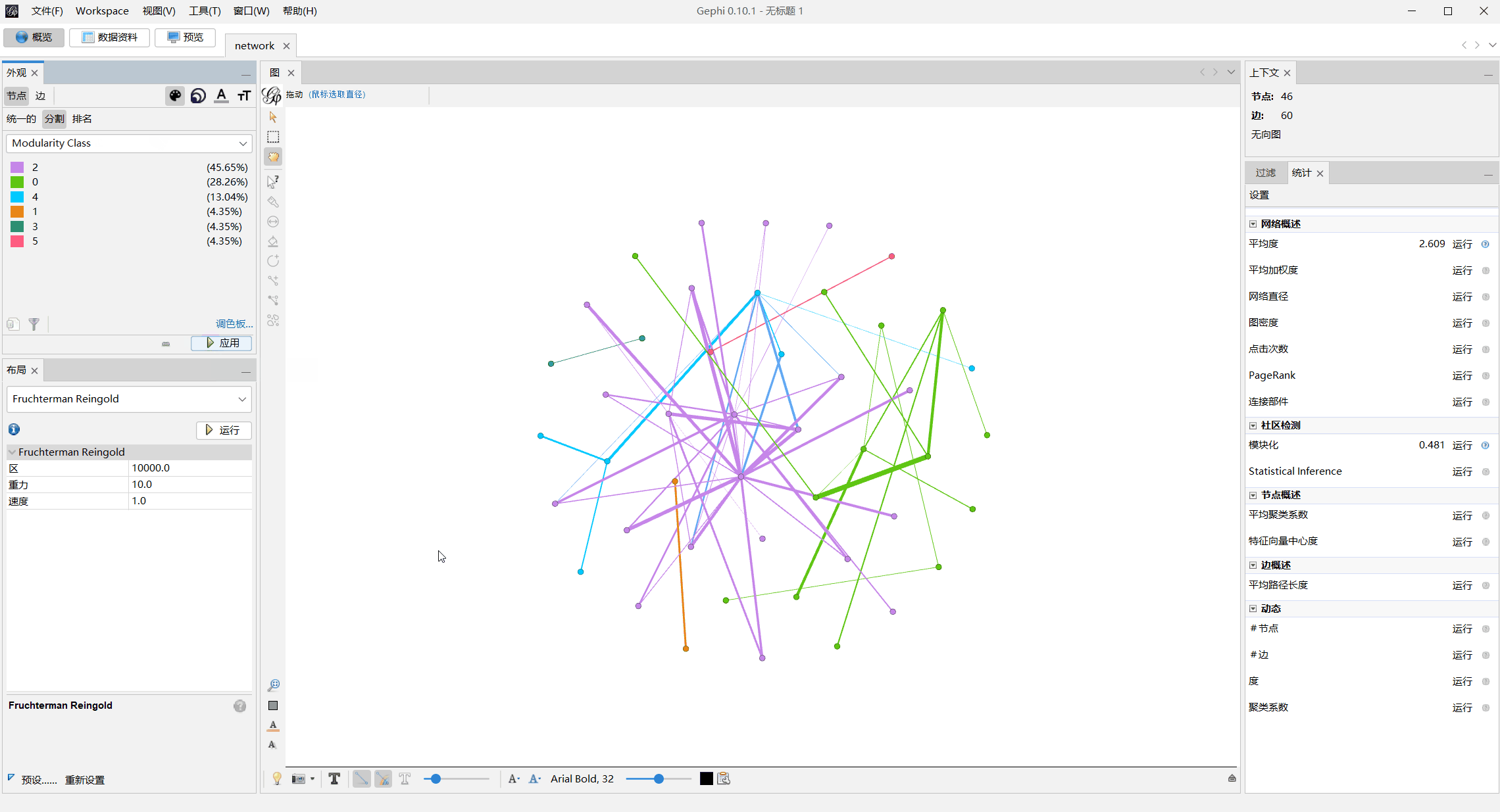
调整节点大小
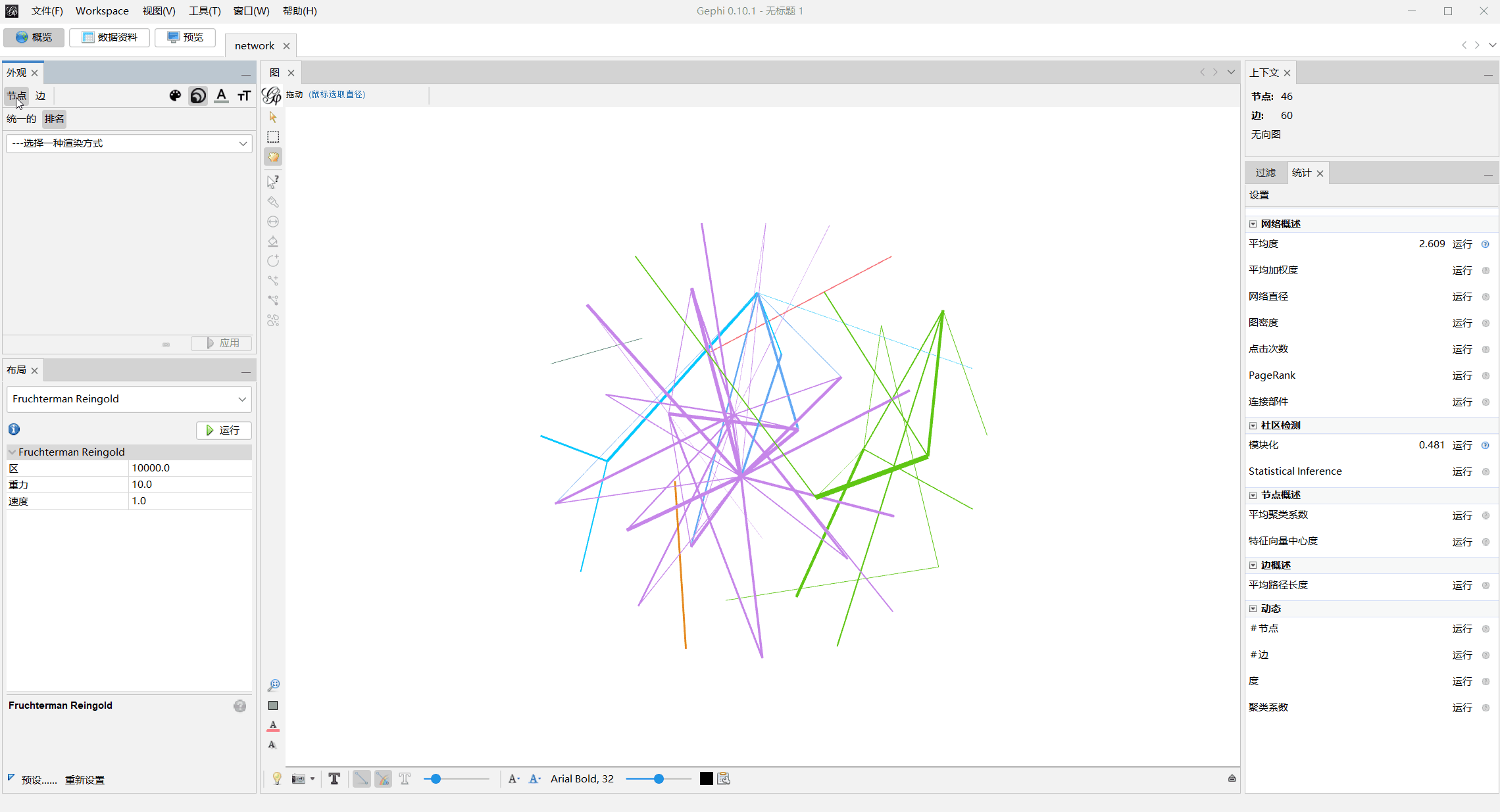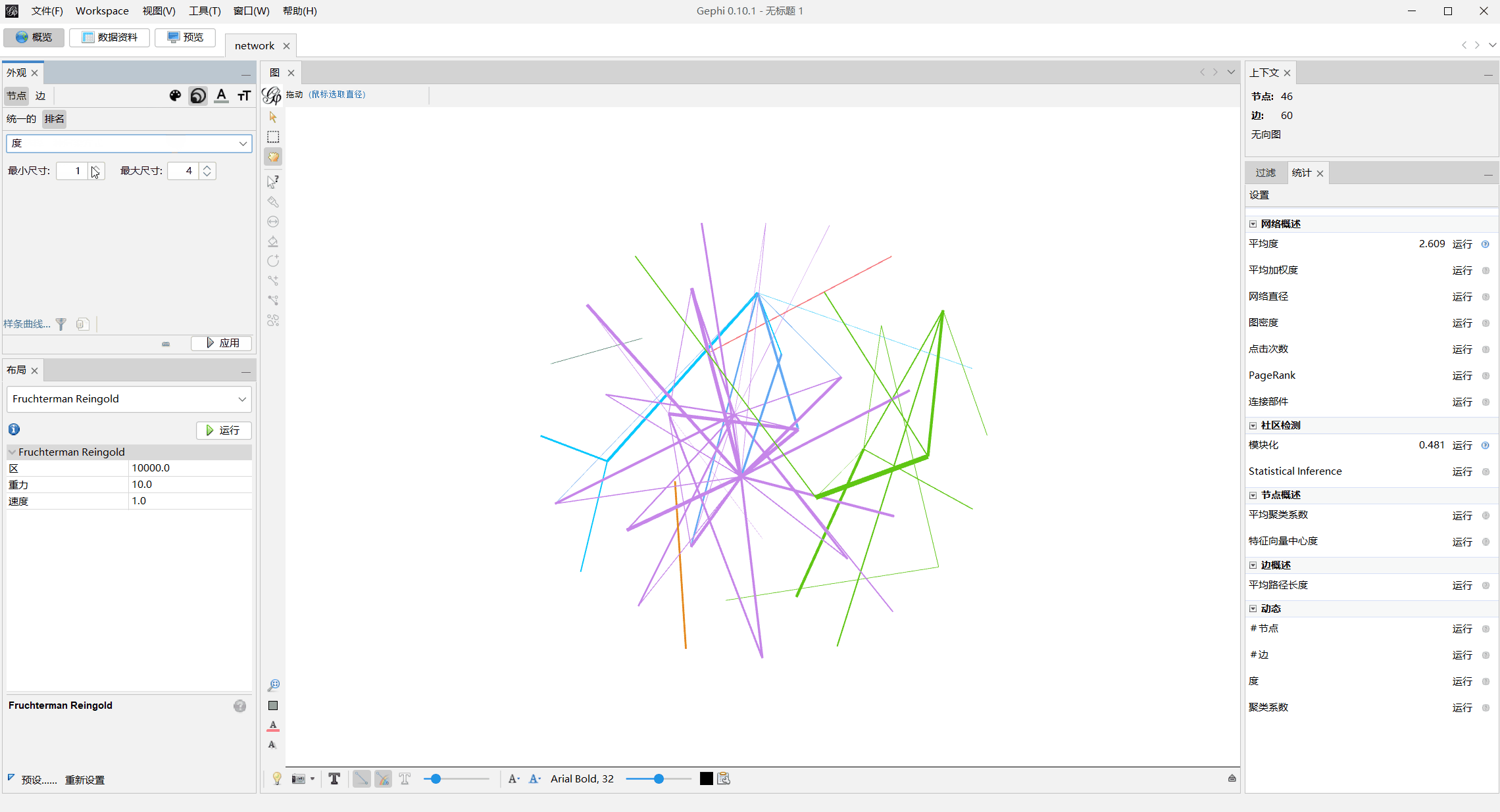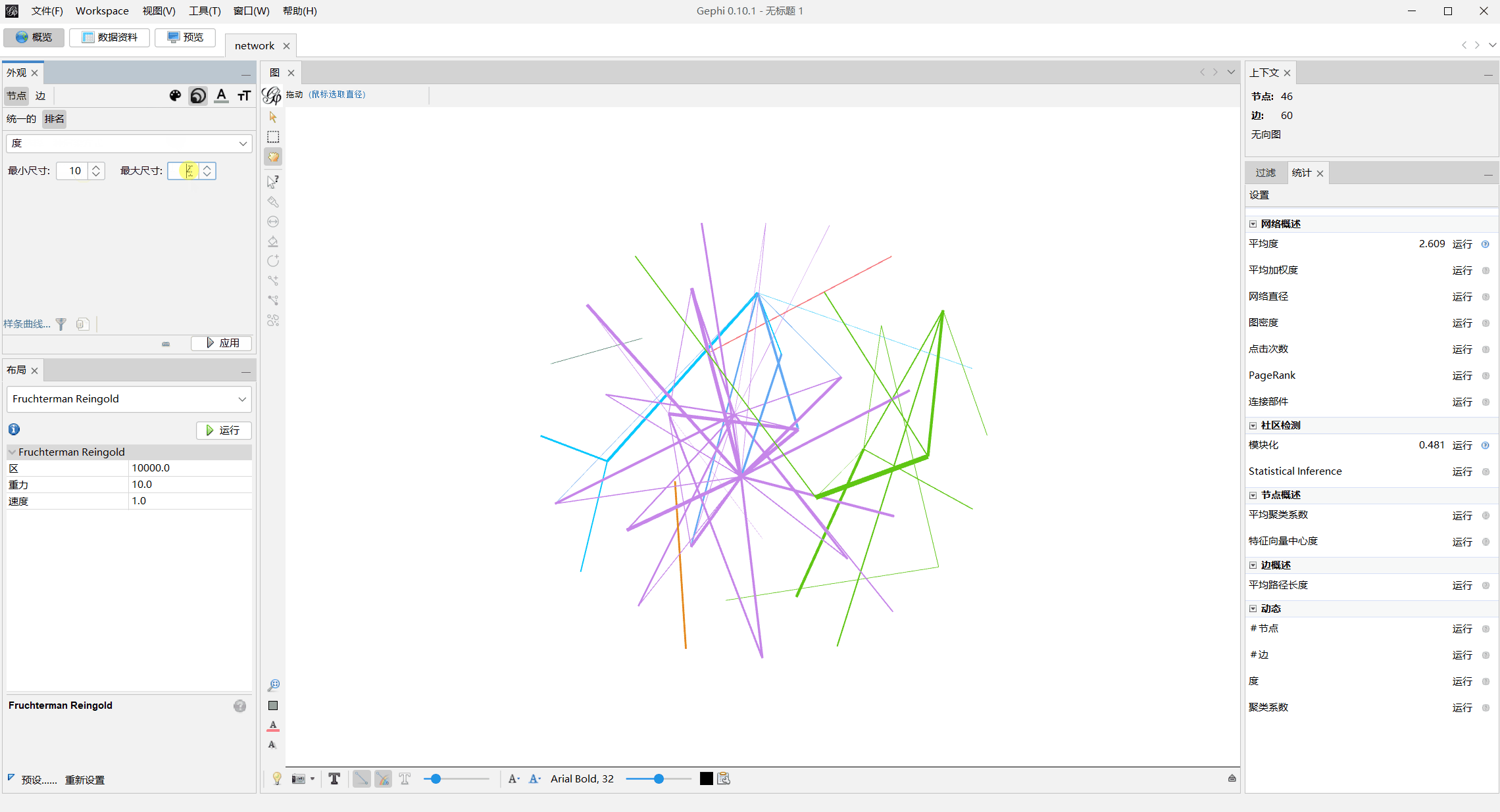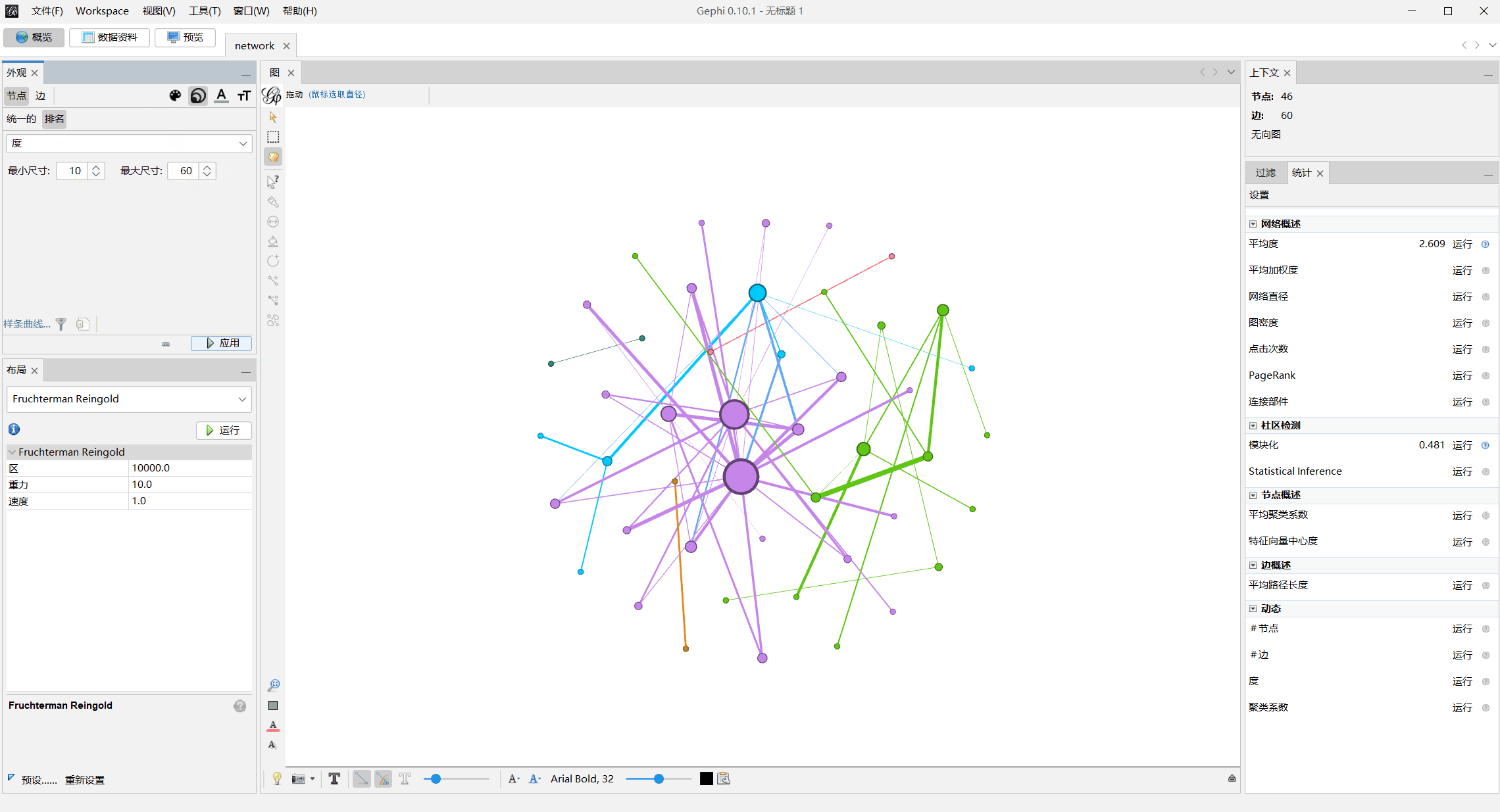
- 添加节点标识,去
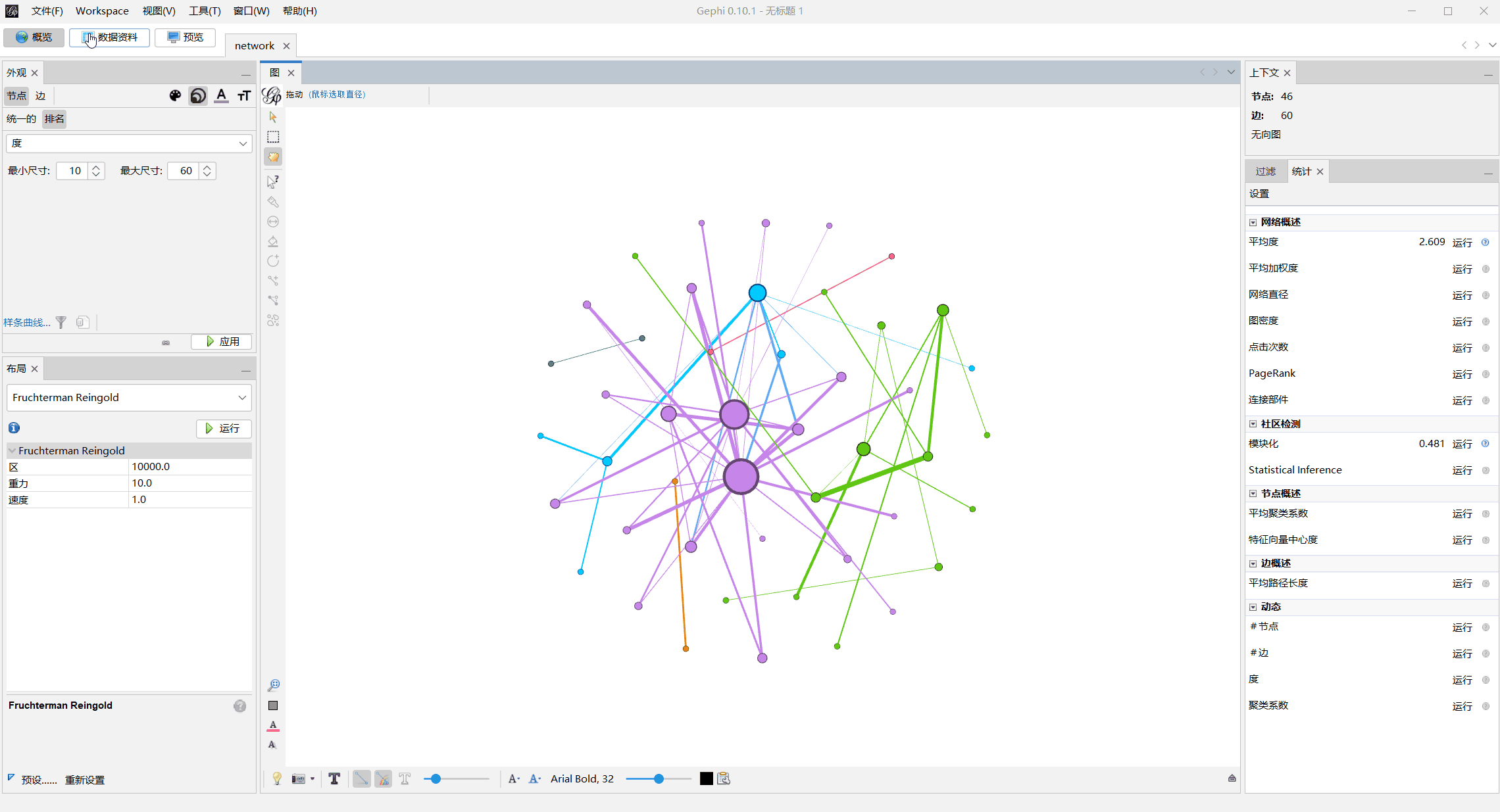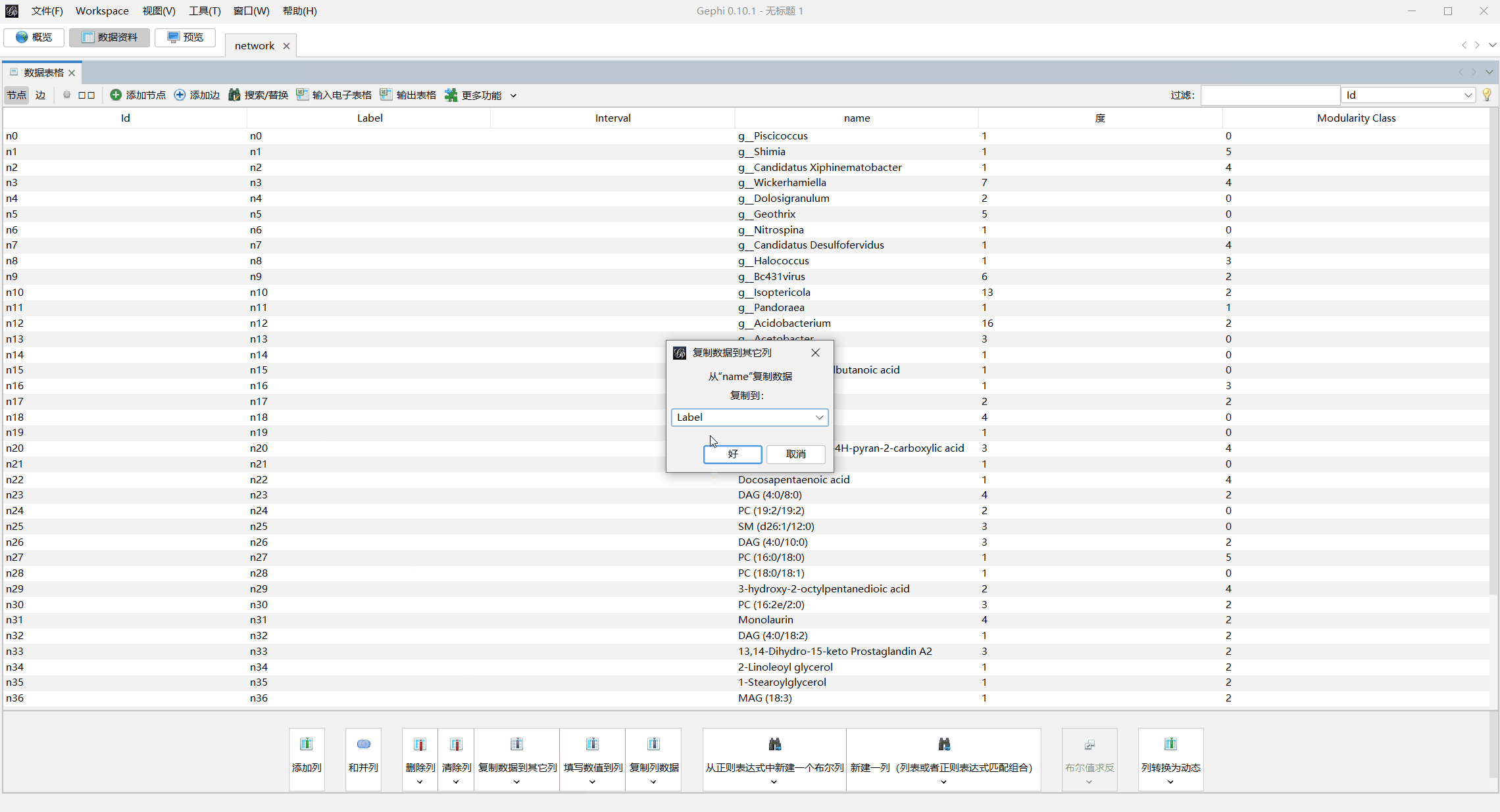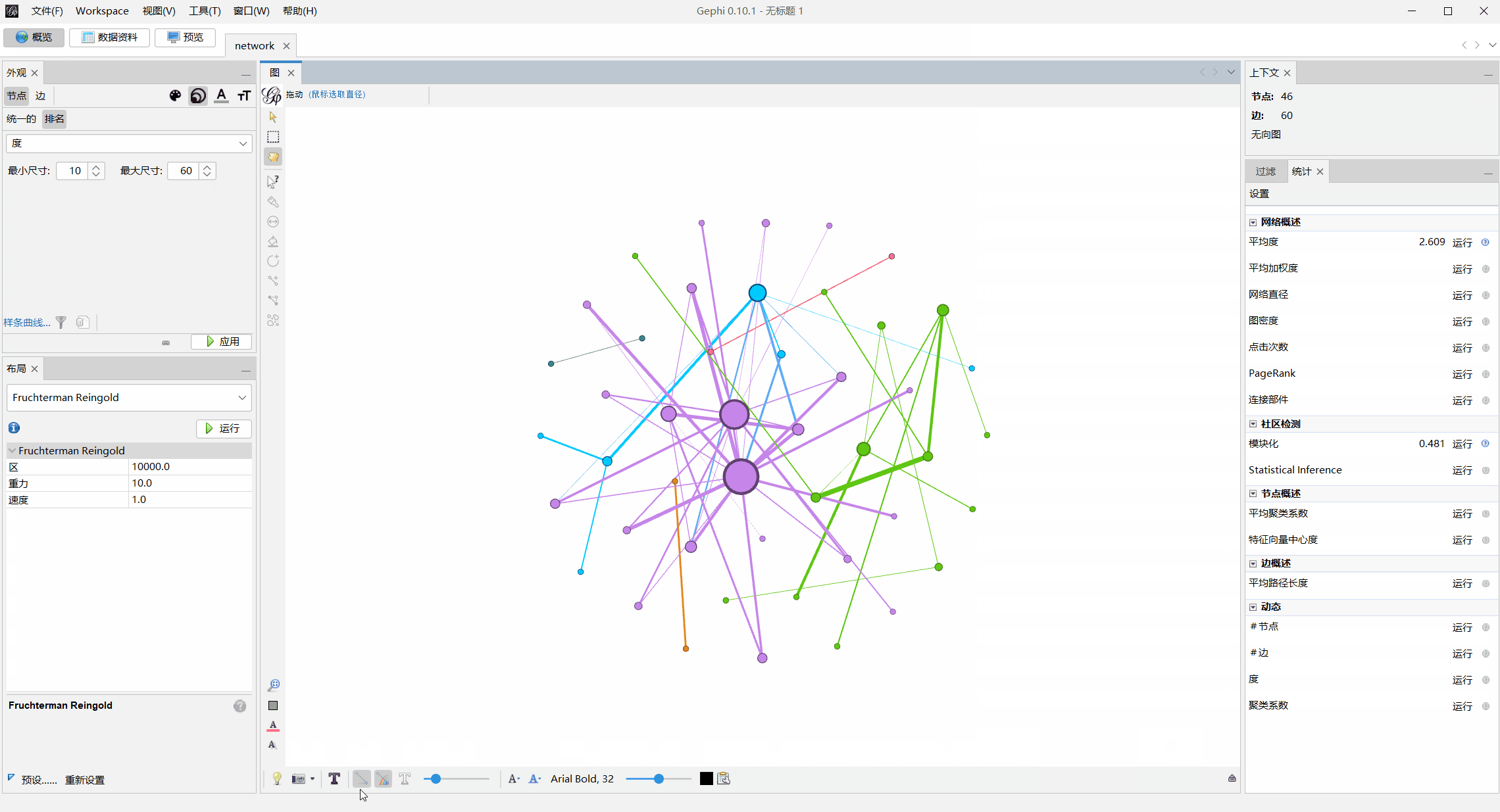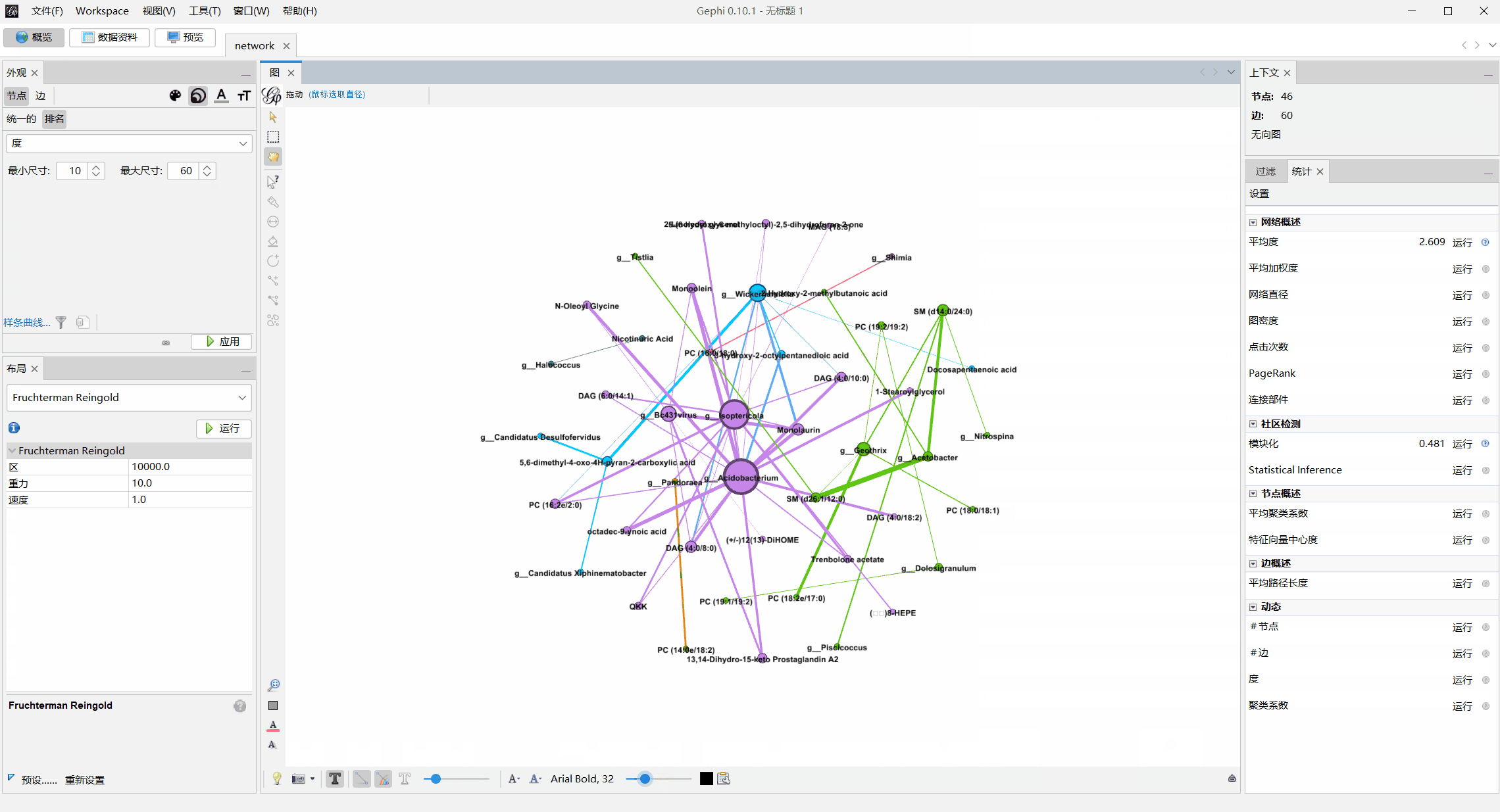
数据资料处替换节点名字
- 预览图,这里根据需要可以自行摸索调整,红框圈出了我调整的部分
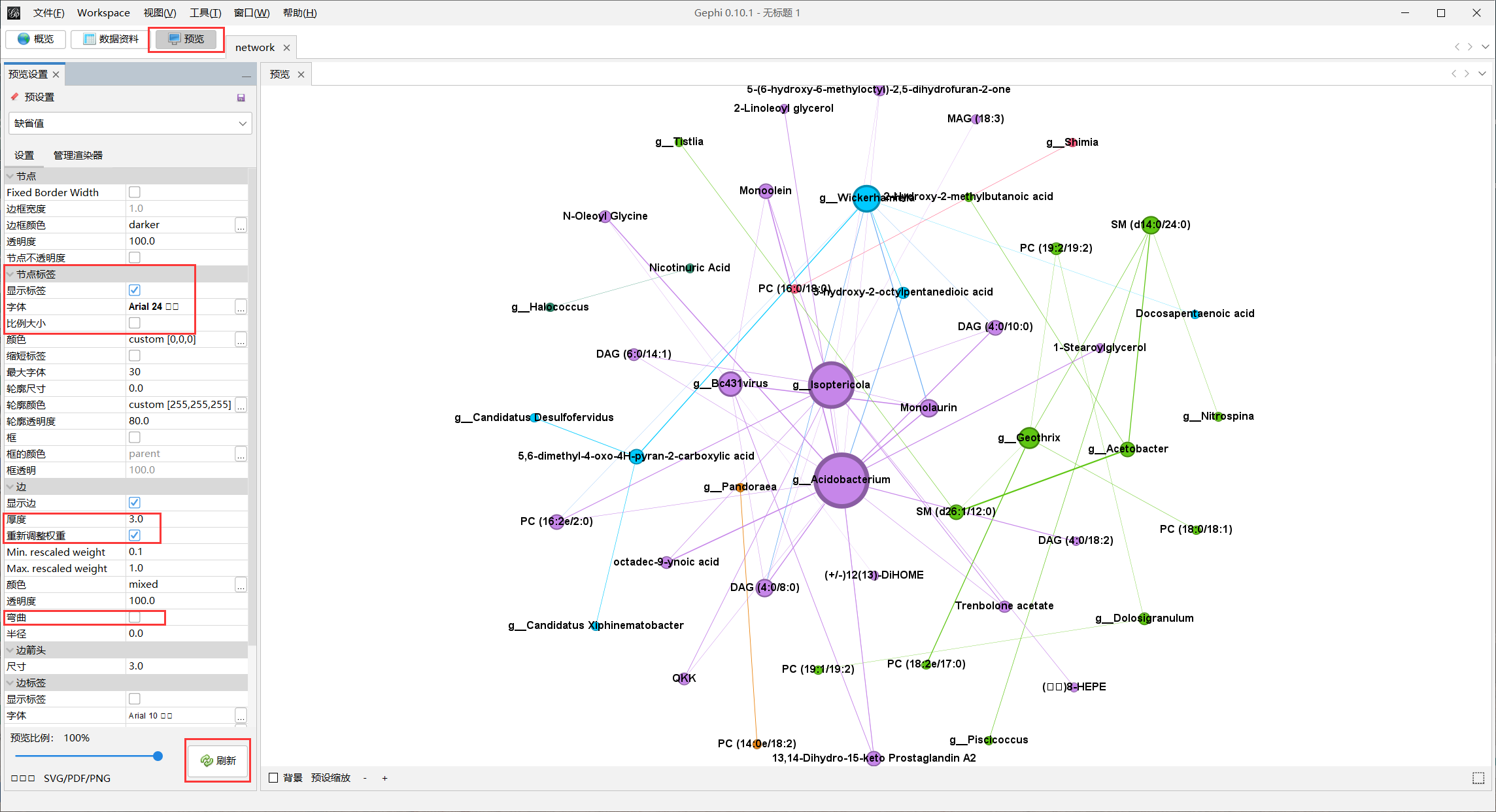
- 导出保存
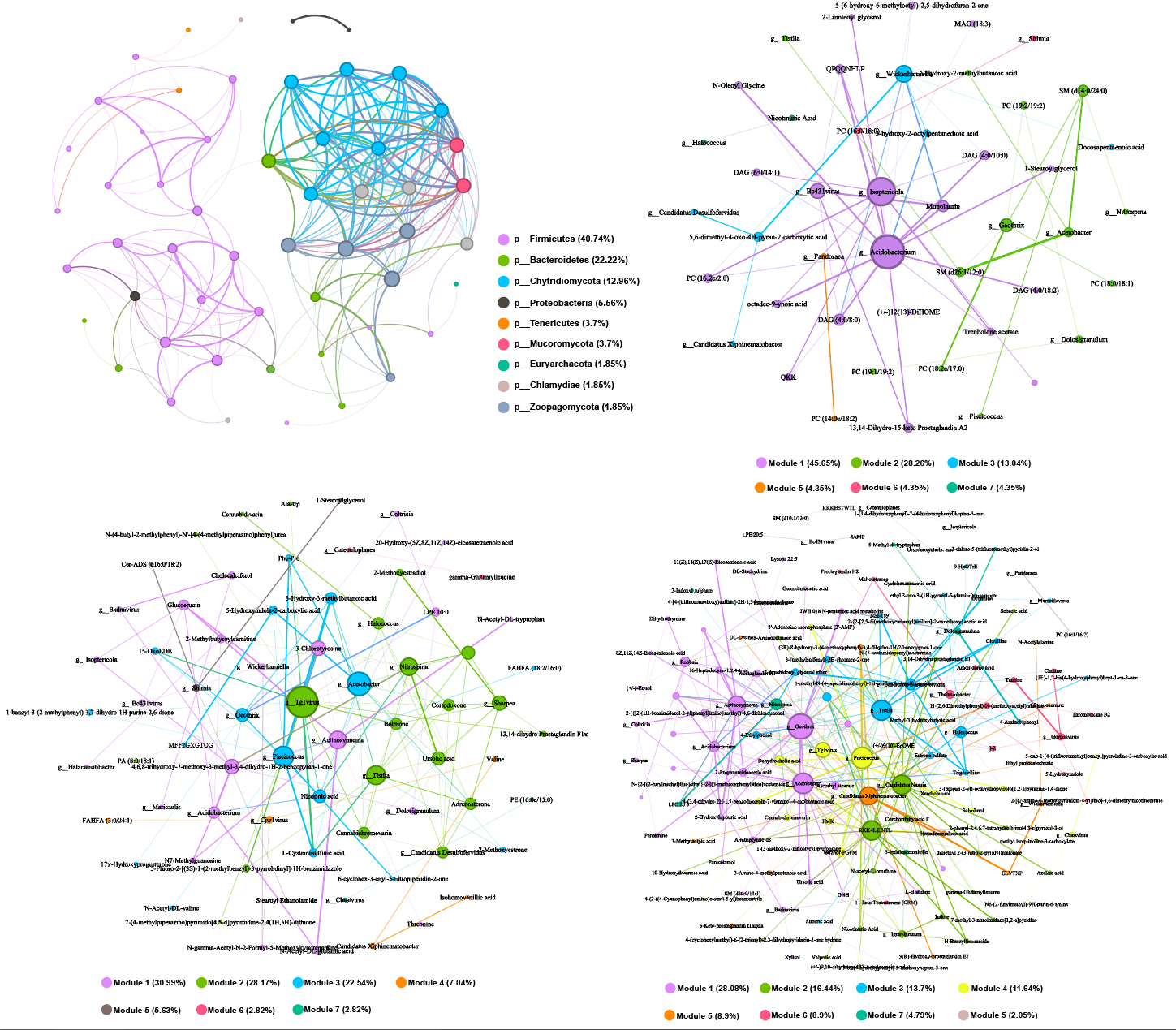
效果展示
结果图细节修改:在AI中嵌入上面导出的pdf格式结果后补充图例细节即可,下方是效果展示
本文链接: https://twocanis.github.io/2023/10/11/%E5%9F%BA%E4%BA%8ER%E5%92%8Cgephi%E5%81%9A%E5%AE%8F%E5%9F%BA%E5%9B%A0%E7%BB%84%E4%B8%8E%E4%BB%A3%E8%B0%A2%E7%BB%84%E7%AD%89%E5%A4%9A%E7%BB%84%E5%AD%A6%E8%81%94%E5%90%88network%E7%9B%B8%E5%85%B3%E6%80%A7%E7%BD%91%E7%BB%9C%E5%9B%BE/
版权声明: 本作品采用 知识共享署名-非商业性使用-相同方式共享 4.0 国际许可协议 进行许可。转载请注明出处!



